Artikel • Bildanalyse in der Onkologie
Machine Learning – fast so gut wie das menschliche Auge?
Machine Learning spielt in der computergestützten Diagnostik eine immer größere Rolle und auch Big Data dringt langsam in die onkologische Bildgebung vor.
Bericht: Mélisande Rouger
Bis sich diese Trends jedoch in der klinischen Routine niederschlagen, kann es noch eine Weile dauern, meint Julia Schnabel, eine deutsche Wissenschaftlerin, die aktuell in England forscht und bei der letzten Jahrestagung der ESMRMB ihre Erkenntnisse präsentierte. Dank Machine Learning-Techniken erkennen Computer heute auch komplexe Bilder fast so gut wie das menschliche Auge. Tools wie etwa das Convolutional Neural Network (CNN oder ConvNet), eine Kategorie der Deep Neural Networks, die zur Bildanalyse eingesetzt wurde, sind für die Segmentierung von Bildern äußerst wichtig geworden.
Analyse der riesigen Datenmengen bleibt eine Herausforderung

Bevor eine akzeptable Bildanalyse jedoch Realität werden kann, gilt es, eine Reihe von Hindernissen zu überwinden, angefangen bei den riesigen Mengen an Daten, die Analysten bewältigen müssen, so Julia Schnabel, Professorin für Computational Imaging am King’s College in London. „In der Bildgebung besteht die größte Herausforderung in der Tatsache, dass wir in 3D oder gar 4D und mit einer Vielzahl von Features arbeiten. Wenn wir Glück haben, sind es Hunderte oder Tausende, aber nicht Millionen von Bildern. Nicht die Anzahl der Bilder ist hier jedoch das Problem, sondern die Größe der Bilddateien.” Das ist nicht das einzige Problem, wie Professor Schnabel weiß, hinzu kommen die immens hohen Kosten und die Mängel in den Trainingsdaten, die unter Umständen, je nach Fachwissen des Auswerters, falsch gekennzeichnet sind.
Machine Learning ist ein ressourcenintensives Unterfangen, denn bestimmte Aufgaben können nur von Spezialisten und Fachärzten ausgeführt werden. „Ich persönlich könnte keinen milchglasartigen Knoten von einem subsoliden Lungenrundherd unterscheiden. Nur Fachärzte und spezielle Radiologen können das“, so Professor Schnabel. Für Krankheiten wie Krebs benötigt daher jedes Bildanalyse-Team die Bestätigung durch einen Pathologen, die häufig nur schwer einzuholen ist. In der Bildgebung des Gehirns muss ein weiteres Hindernis überwunden werden: Es existieren verschiedene Protokolle, das heißt, die gleiche Krankheit kann je nach Protokoll und Patient unterschiedlich dargestellt werden. „Lage und Größe einer Läsion können sich stark unterscheiden, und eine Pathologie kann als klar abgegrenzter, diffuser oder infiltrierter Herd auftreten“, erklärt Professor Schnabel.
In den Trainings- oder Testdaten gibt es häufig ein Ungleichgewicht aufgrund der unterschiedlichen Anteile von gesunden versus pathologischen Fällen
Julia Schnabel
Die Expertin gibt praktische Empfehlungen für die Arbeit mit dem CNN und betont, die Menge der Daten, die aus einem Bild herausgelesen werden kann, hängt von der Größe des rezeptiven Felds ab: „Die Größe der verwendeten Patches ist wichtig, denn ein großes rezeptives Feld erhöht die Anforderungen an die Rechnerleistung und den Speicherplatz. Max-Pooling mag zu einem Verlust der räumlichen Auflösung führen, aber kleine Patches sind sehr rauschanfällig.“ Als Lösung schlägt Schnabel einen Multiskalen-Ansatz vor, d. h. kleine Patches werden mit kleineren Filtern und größere Patches mit größeren Filtern verwendet. Am Ende erst werden alle zusammengeführt.
Die onkologische Bildanalyse bringt ihre ganz eigenen Probleme mit sich. Segmentierung auf der Basis von Machine Learning verliert in klinischen Szenarien häufig an Aussagekraft. Dies ist den Unterschieden zwischen Trainings- und Testdaten zuzuschreiben, die sich wiederum aus den Unterschieden in der Hardware, den Scannerprotokollen und den Sequenzen ergeben, so Schnabel: „In den Trainings- oder Testdaten gibt es häufig ein Ungleichgewicht aufgrund der unterschiedlichen Anteile von gesunden versus pathologischen Fällen, der Variabilität zwischen einzelnen Patienten und Krankheitsbildern, auch innerhalb desselben Patienten. Leberläsionen sind zum Beispiel meist Sekundärtumore, die von einen Primärtumor in einem anderen Organ, etwa dem Dickdarm, hervorgerufen werden.“ Daher ist die Auswahl der geeigneten Netzwerkarchitektur von eminenter Bedeutung. Derzeit werden in der Literatur drei interessante Modelle diskutiert: DeepMedic, FCN (im Deep Learning Toolkit) und U-Net, das seinen Namen seiner U-Form verdankt. „Alle drei Netzwerke verfolgen unterschiedliche Ansätze, und jeder Ansatz hat seine Vor- und Nachteile“, erläutert Professor Schnabel.
Das Netzwerk-Ensemble übernimmt
Obzwar alle drei Netzwerke auf CNN-Basis arbeiten und auch gute Ergebnisse erzielen, gibt es eine Menge Meta-Parameter, mehr als Input-Fälle, und sowohl Architektur als auch Konfiguration beeinflussen Performance und Verhalten. Der Nachteil aller Ansätze: Die ausgewählten Modelle und Parameter sind unter Umständen für andere Daten und Anwendungen wenig geeignet. Das wiederum, so Professor Schnabel, könne dazu führen, dass „Ergebnisse und Schlussfolgerungen stark verzerrt sind.“ Der Einsatz eines sog. Netzwerk-Ensembles wie EMMA (Ensemble of Multiple Models & Architectures) kann eventuell Abhilfe schaffen, denn weder wird die Performance hier durch suboptimale Konfiguration beeinträchtigt, noch wird das Verhalten durch Architektur und Konfiguration verzerrt.
Zur Segmentierung von Darmkrebs-Bildern, die mit dynamischer Kontrast-MRT (DCE MRI) akquiriert wurden, extrahierten Schnabel und ihr Team mithilfe der sog. Principal Component Analysis die Variabilität normalisierter Signalintensitätskurven aus dem Datensatz. „Das ist im Grunde eine einfache Technik. Wir betrachten die mittlere Signalintensität der Kurven in einem übersegmentierten Ansatz, sogenannte Superpixel oder Supervoxel, in 3D. Dann klassifizierten wir Perfusionssupervoxel in nicht gesehenen Fällen mithilfe von Support Vector Machines. So erhalten wir die Tumorsegmentierungen.“ Die Ergebnisse dieser automatisierten Methode liegen innerhalb der Urteilerübereinstimmung von zwei Experten, jedoch wurde ein Korrekturschritt notwendig, als die Segmentierungsmaske von der T2W-Sequenz auf die T1W-Sequenz in der DCE MRI übertragen wurde, so Schnabel.
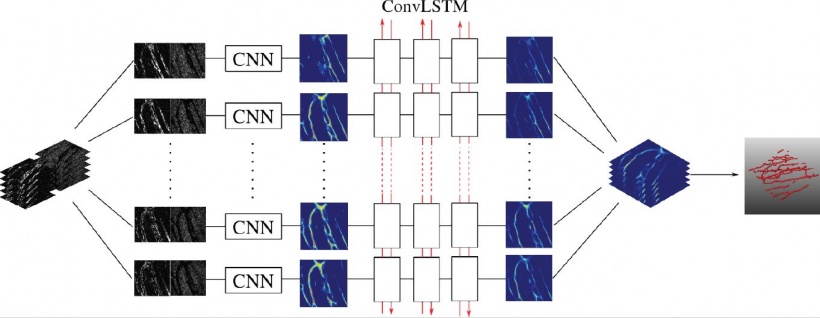
Für eine bessere Tumor-Segmentierung
„Tumore haben ganz unterschiedliche Formen, das heißt, wir brauchen in gewissem Umfang anatomischen Kontext. Dazu haben wir eine graphische Darstellung der umgebenden Anatomie entwickelt. Wir verbessern die Segmentierung, wenn wir sowohl lokale als auch globale Beziehungen beachten. Wenn wir wissen, wo die Blase liegt und wie das Lumen aussieht, können wir sie in ein Modell der Teile von Teilen integrieren. Wenn wir dann diese Teile von Teilen klassifizieren, reduzieren wir die Anzahl der falsch-positiven Befunde und erhalten letztendlich eine genauere Tumorsegmentierung“, so Professor Schnabel.
Schnabel und ihre Kollegen haben mit der sog. Tumor-Parzellierung auch eine Segmentierung innerhalb eines Tumors durchgeführt. So konnten sie aus den DCE MRI-Scans lokal relevante zusammenhängende Perfusionssubregionen extrahieren. Zehn weibliche CBA-Mäuse mit subkutan implantierten CaNT-Tumoren wurden über einen Zeitraum von acht Tagen gescannt, um das Tumorwachstum und das Clustering der Perfusionssupervoxel zu beobachten. Die Visualisierung der Gefäße eröffnete sogar noch einen tieferen Blick in den Tumor, berichtet Professor Schnabel: „Wir haben konfokale 3D-Fluoreszenzmikroskopie eingesetzt, eine Bildgebungstechnologie mit stark anisotropen Voxeln, die nur einige Mikronen groß sind. Endothel- und Tumorzellen wurden mit Fluoreszenz gekennzeichnet und etwa 60 Schichten wurden auf der z-Achse generiert. Die Vaskulatur war noch bis zu 30 Schichten unterhalb der Oberfläche erkennbar.“
In der Bildanalyse von Lungenkrebs liegt der Schwerpunkt auf der Erkennung von Lungenknoten oder Lymphknoten im Lungen-CT. Deep Learning ersetzt heute schon weitgehend konventionelle CADe- und CADx-Methoden, die auf der Analyse von Texturen, eigens definierten Features und einfacher Klassifizierung beruhen. Laut Schnabel sind die Nachweisquoten mit Deep Learning im Allgemeinen sehr hoch, so dass man sich heute auf die Reduzierung der falsch-positiven Befunde konzentriert.
Big Data oder Dense Data?
In der onkologischen Bildgebung und Bildanalyse ist Machine Learning ein vielversprechendes Werkzeug. Die Herausforderung besteht nach wie vor darin, die richtigen Modell-Parameter für gute Schätzungen und Verallgemeinerungen zu finden. „Wir haben multidimensionale und multimodale Datensets, aber das ist weniger Big Data als Dense Data – dichte Daten. Was uns wirklich helfen würde“, so Professor Schnabel abschließend, „sind Kohortenstudien, die große Mengen an Daten sammeln.“
Profil:
Julia Schnabel ist seit Juli 2015 Inhaberin des Lehrstuhls „Computational Imaging“ der Abteilung Imaging Sciences & Biomedical Engineering des King's College London und Direktorin des EPSRC Centre for Doctoral Training in Medical Imaging, einem gemeinsamen Projekt des King’s College London und des Imperial College London. Darüber hinaus ist sie Visiting Professor in Engineering Science an der Universität Oxford.
17.04.2018