Bild: C. Gatsogiannis / MPI für molekulare Physiologie
News • Proteinkomplex ClpX-ClpP
Erreger-Schwachstelle liefert "Munition" für neue Therapien
Noch immer sind Antibiotika die wichtigste Waffe zur Bekämpfung bakterieller Infektionen. Jedoch geht der Medizin aufgrund von immer häufiger vorkommenden Resistenzen die „Munition“ aus.
Ein Forschungsteam hat nun die Struktur des proteinabbauenden Komplexes ClpX-ClpP aufgeklärt. Dies ist ein Schlüssel zur Entwicklung innovativer Antibiotika, die auf den Abbauprozess von defekten Proteinen in Bakterien abzielen. Fast 700.000 Menschen erkranken in der EU jährlich an Infektionen durch antibiotikaresistente Erreger, circa 33.000 von ihnen sterben. Trotz dieser enormen und weltweit zunehmenden Gefahr wurden in den letzten Jahrzehnten nur wenige neue Antibiotika entwickelt und zugelassen. Eine Verbesserung ist nicht in Sicht. Deshalb ist es dringend notwendig, neue Angriffspunkte in krankheitserregenden Bakterien zu finden und neuartige Antibiotika zu entwickeln, die diese Schwachstellen ausnutzen.
Dieser Artikel könnte Sie auch interessieren

Artikel • Differenzieren und selektieren
Mythen und Wahrheiten über Antibiotika, Antiseptika und Impfungen
62 Prozent der Deutschen fürchten sich vor Antibiotikaresistenzen, das ergab jüngst eine Umfrage des Bundesinstituts für Risikobewertung. Gerade auch Patienten, die mit multiresistenten Erregern besiedelt sind, leben häufig in Angst. Dabei beruhen viele Ängste auf Missverständnissen.
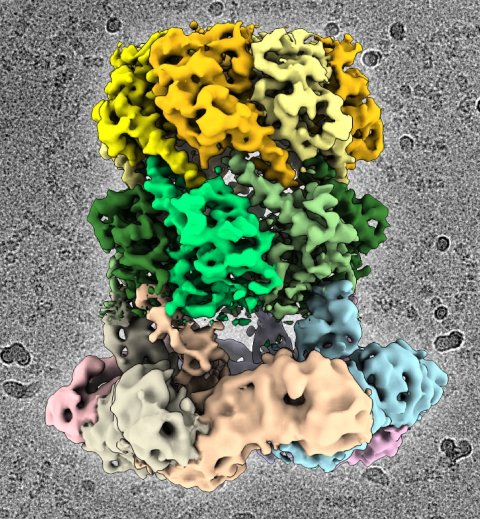
Bild: C. Gatsogiannis / MPI für molekulare Physiologie
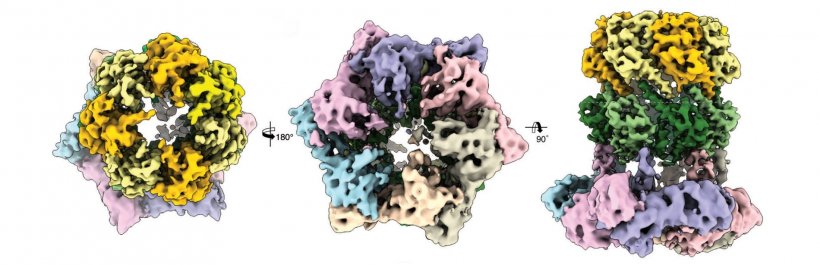
Ein vielversprechendes Ziel für antibakterielle Therapien ist das proteinabbauende Enzym ClpP. Es spielt zum einen eine wichtige Rolle im bakteriellen Stoffwechsel und sorgt für den kontrollierten Abbau defekter Proteine. Dazu benötigt es jedoch das Protein ClpX als Starthilfe. Im Komplex mit ClpP erkennt ClpX Proteine die abgebaut werden sollen, entfaltet sie und leitet diese dann in seine fassartige Abbaukammer.
Wissenschaftler der Gruppen um Prof. Stephan Sieber, Technische Universität München (TUM) und Prof. Stefan Raunser, Direktor am Max-Planck-Institut für molekulare Physiologe in Dortmund, haben nun erstmals die dreidimensionale Struktur des proteinabbauenden Komplexes ClpX-ClpP aufgeklärt und damit eine wichtige Basis für zukünftige pharmakologische Anwendungen geschaffen.
Eine neue Klasse potentieller Antibiotika, die sogenannten Acyldepsipeptide (ADEP) bewirken einen unkontrollierten Abbau durch ClpP auch ohne die Unterstützung von ClpX. Dadurch werden lebenswichtige Proteine zerstört – mit tödlichen Folgen für die Bakterien. Dieser einzigartige Wirkmechanismus hat ein beträchtliches Innovationspotential im Kampf gegen krankmachende Bakterien. Denn während gängige Antibiotika durch die Hemmung lebenswichtiger Prozesse wirken, wird in diesem Fall der antibakterielle Effekt durch die Aktivierung eines Vorgangs erzielt.
Dieser Artikel könnte Sie auch interessieren

Artikel • Medikamentenentwicklung
Unterstützung vom anderen Ende der Welt
Im Kampf gegen resistente Keime haben sich Partner zusammengetan, die kaum weiter voneinander entfernt sein könnten – und doch eine Menge Gemeinsamkeiten haben.
Neben dem Abbau von defekten Proteinen ist ClpP auch ein entscheidender Regulator bei der Produktion eines Arsenals bakterieller Gifte, die maßgeblich verantwortlich sind für die krankmachende Wirkung vieler Erreger. An der TU München forscht die Gruppe um Prof. Stephan Sieber seit Jahren erfolgreich an der Protease ClpP und hat bereits eine Vielzahl potenter Hemmstoffe gegen ClpP und ClpX entwickelt, die die Produktion von bakteriellen Giften stoppen und sie damit quasi entwaffnen können. Dóra Balogh gelang es nun, den ClpX-ClpP Komplex herzustellen und zu stabilisieren.
Im Detail konnte die Struktur des ClpX-ClpP Komplexes aber bisher noch nicht aufgeklärt werden. Dr. Christos Gatsogiannis, Mitarbeiter in der Gruppe um Prof. Stefan Raunser am MPI für molekulare Physiologe, gelang dies nun mittels Kryo-Elektronenmikroskopie. Mit dieser Technik konnte das Team zeigen, dass ADEP und ClpX zwar an derselben Stelle an ClpP andocken, aber auf unterschiedliche Weise den Prozess des Proteinabbaus steuern. Während ClpX zu keiner Veränderung in der Struktur von ClpP führt, verursacht ADEP eine nicht vorgesehene Öffnung der Protease. Dadurch werden auch intakte Proteine unkontrolliert und ohne die Unterstützung von ClpX abgebaut.
Die Aufklärung dieses Mechanismus durch die Forscherteams aus Dortmund und München ist ein Meilenstein auf dem Weg zur Entwicklung innovativer antibiotischer Substanzen, die ClpP als Angriffsziel haben.
Quelle: Technische Universität München (TUM)
05.10.2019