News • Epigenetische Analyse
Dem Tumor den richtigen Namen geben
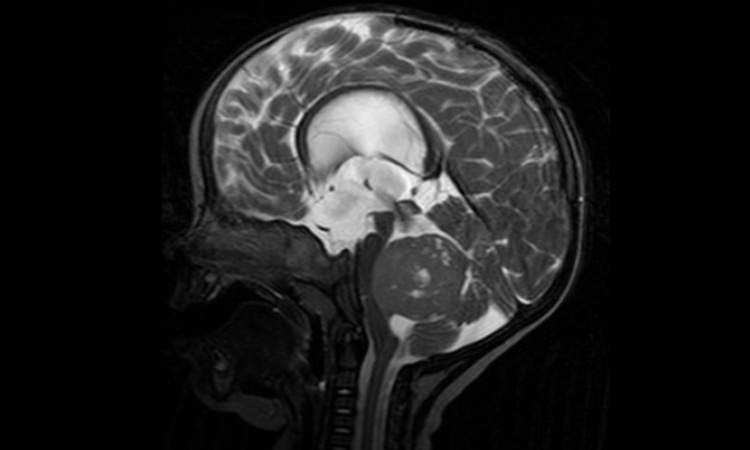
Wissenschaftler des „Hopp-Kindertumorzentrums am NCT Heidelberg (KiTZ)“ und der Abteilung Neuropathologie am Universitätsklinikum Heidelberg haben die Klassifikation von Tumoren des Zentralen Nervensystems (ZNS) entscheidend verbessert
Damit Krebs des zentralen Nervensystems erfolgreich behandelt werden kann, ist es wichtig, die molekularen Eigenschaften der Tumoren genau zu kennen und den Tumoren damit „den richtigen Namen zu geben“. Zurzeit lassen sich über Gewebemerkmale rund 100 verschiedene Arten von ZNS-Tumoren unterscheiden, die ganz unterschiedlich auf Strahlen- und Chemotherapien ansprechen. Mitunter werden molekulardiagnostische Methoden eingesetzt, um den Tumor näher zu klassifizieren, zum Beispiel über bestimmte Genmutationen. Dennoch ist die Variabilität groß, was die Standardisierung von Diagnoseverfahren erschwert.
Wir haben computerbasierte Algorithmen entwickelt, die 82 verschiedene Arten von ZNS-Tumoren anhand ihrer Methylierungsmuster zuverlässig unterscheiden können
David Capper
Um die Diagnostik von ZNS-Tumoren zu verbessern, entwickelte das Team um Professor Stefan Pfister, KiTZ-Direktor und Abteilungsleiter „Pädiatrische Neuroonkologie“ am DKFZ, zusammen mit Kollegen der Abteilung Neuropathologie am Heidelberger Universitätsklinikum unter der Leitung von Professor Andreas von Deimling, ein neues computerbasiertes Verfahren: „Wir hoffen, die diagnostische Treffsicherheit bei ZNS-Tumoren mit unserer neuen, molekularen Klassifizierungsmethode zu verbessern und damit auch die Erfolgsaussichten der anschließenden Therapie zu erhöhen“, erklärt von Deimling.
Die Forscher analysierten bestimmte chemische Markierungen im Erbgut von Tumoren, sogenannte DNA-Methylierungen. Verschiedene Zelltypen weisen charakteristische Muster an DNA-Methylierungen in ihrem Erbgut auf, die wiederum Rückschlüsse auf die zelluläre Herkunft des Tumors zulassen. „Wir haben computerbasierte Algorithmen entwickelt, die 82 verschiedene Arten von ZNS-Tumoren anhand ihrer Methylierungsmuster zuverlässig unterscheiden können“, sagt Professor David Capper, einer der vier Erstautoren der Studie, der inzwischen als Fakultätsmitglied des DKTK-Standortes Berlin eine Professur für molekulare Neuropathologie an der Charité – Universitätsmedizin Berlin angenommen hat. „Gerade bei Tumoren, die wir durch die reine Betrachtung unter dem Mikroskop nicht ohne weiteres einer diagnostischen Kategorie zuordnen können, hilft die Methylierungsanalyse häufig, eine eindeutige Diagnose zu stellen. Die Analyse der insgesamt circa 2800 Tumor-Referenzproben ermöglichte zudem die Abgrenzung bestimmter Tumor-Subgruppen, die in den bisher gängigen Klassifizierungen noch gar nicht enthalten sind.“
Einsatz in der Klinik
Um zu testen, ob sich die Methode für den Einsatz in der klinischen Routinediagnostik eignet, analysierten die Wissenschaftler mehr als 1100 weitere Tumorproben. In rund 12 Prozent der Tumoren konnten sie mithilfe der Methylierungsmuster die ursprüngliche Diagnose korrigieren. Weiterführende molekulardiagnostische Untersuchungen zeigten in fast allen Fällen, in denen dies möglich war, dass die molekulare Zuordnung die Tumoren sogar besser charakterisierte als die ursprüngliche mikroskopische Diagnose. „Wir sind überzeugt, dass sich unsere neue Methode gut für den Einsatz in der Klinik eignet“, sagt Pfister und ergänzt: „Wir haben unser Klassifizierungssystem online zugänglich gemacht, damit Forscher ihre Daten auf unserer Plattform analysieren können.“ Durch die Informationen, die auf diese Weise gleichzeitig eingehen, sollen vor allem seltene Krebserkrankungen in Zukunft eindeutig diagnostiziert und damit besser behandelt werden können.
Die Online-Plattform zur DNA-Methylierungsanalyse ist unter www.molecularneuropathology.org verfügbar.
Quelle: Universitätsklinikum Heidelberg
15.03.2018