E. coli
Wie pathogene Darmbakterien die Magensäure überleben
Krankheitserregende Darmbakterien aktivieren Schutzmechanismen, um im sauren Milieu des Magens zu überleben. Forscher des Nationalen Forschungsschwerpunktes (NFS) «TransCure» der Universität Bern haben nun die Struktur des zentralen Proteins eines bakteriellen Säure-Resistenz-Systems entschlüsseln können. Dadurch werden wichtige Einblicke in die «Überlebenstricks» von Darmbakterien gewonnen.
Durch die Nahrung aufgenommene krankheitserregende Darmbakterien werden den extrem sauren Bedingungen des Magens ausgesetzt. Um dieses unwirtliche Milieu zu überleben und anschliessend in den menschlichen Darm zu gelangen, aktivieren Bakterien verschiedene Überlebensmechanismen.
Das am besten charakterisierte bakterielle Säure-Resistenz-System stammt vom Darmbakterium Escherichia coli (E. coli). Bestimmte Stämme von E. coli sind häufig Verursacher von menschlichen Darmerkrankungen. Für das Überleben von E. coli im Magen spielt ein bestimmtes Protein eine bedeutende Rolle. Wie es aber auf molekularer Ebene die Darmbakterien vor der Magensäure schützt, war bislang nicht klar.
Nun ist es Hüseyin Ilgü und Jean-Marc Jeckelmann aus der Gruppe von Prof. Dimitrios Fotiadis vom Institut für Biochemie und Molekulare Medizin der Universität Bern und dem NFS «TransCure» gelungen, die Struktur dieses Proteins zu entschlüsseln und offene Fragen zu seiner Funktionsweise zu beantworten.
Transportprotein zentral für das Überleben
Werden E. coli-Zellen sauren Bedingungen – das heisst tiefen pH Werten – ausgesetzt, können Protonen über die Zellwände eindringen und das Zellinnere ansäuern – dies führt zum Absterben der Zellen. Um dies zu vermeiden, aktivieren Bakterien Mechanismen, die ein Überleben bei tiefem pH-Wert sicherstellen. Säure-Resistenz-Systeme sorgen dafür, dass im Zellinneren eine pH-Untergrenze nicht unterschritten wird, um das Überleben des Bakteriums zu ermöglichen.
So werden bei der Umwandlung der Aminosäure L-Arginin in das Molekül Agmatin Protonen gebunden und somit aus dem bakteriellen Zellinneren entfernt. Die Abnahme der Protonenkonzentration in der Zelle hat die Erhöhung und Stabilisierung des pH-Wertes im Zellinneren zur Folge. Um frisches L-Arginin ein- und Agmatin auszuschleusen, verwendet E. coli das Transportprotein AdiC. Transportproteine befinden sich in den Wänden von Zellen und agieren als «Türsteher», indem sie nur bestimmte Moleküle hinein- und herauslassen.
Vom Verständnis der Funktion zum Arzneistoff
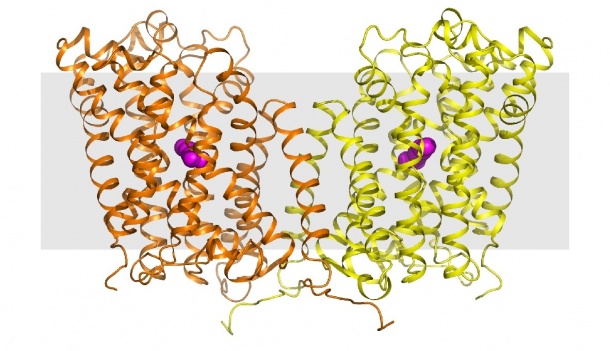
Der molekulare Transportmechanismus von AdiC ist unklar, ebenso wie die Erkennung und Bindung der Substrate L-Arginin und Agmatin durch den Transporter. Um dies klären zu können, hat die Gruppe um Dimitrios Fotiadis Kristalle von AdiC-Transportern gezüchtet und diese für die Entschlüsselung der atomaren Struktur mittels Röntgenkristallographie verwendet. Dank des äusserst präzisen atomaren Modells von AdiC in An- und Abwesenheit von gebundenem Agmatin konnten nun zum ersten Mal wichtige offene Fragen zur molekularen Erkennung und Bindung von L-Arginin und Agmatin an das Protein beantwortet werden.
Die Proteinstuktur dient zudem als guter Ausgangspunkt für das Verständnis des Transportmechanismus von L-Arginin und Agmatin durch die Membran. «Die gewonnenen strukturellen und funktionellen Erkenntnisse von AdiC sind auch für die Entwicklung von Arzneistoffen innerhalb des Berner NFS «TransCure» wertvoll» sagt Fotiadis. So wird nun die Struktur von AdiC bei der Suche nach Hemmern von menschlichen Transportern eingesetzt, welche mit AdiC verwandt sind und in bestimmten Krebszellen überexprimiert werden.
Publikation:
Hüseyin Ilgü, Jean-Marc Jeckelmann, Vytautas Gapsys, Zöhre Ucurum, Bert L. de Groot, Dimitrios Fotiadis: Insights into the molecular basis for substrate binding and specificity of the wild-type L-arginine/agmatine antiporter AdiC, PNAS, 29. Juli 2016, doi: 10.1073/pnas.1605442113
Quelle: Universität Bern
29.09.2016