Copyright: Karin Kaiser / MHH
News • Personalisierte Medizin
Leukämie bei Kindern: Bessere Diagnostik durch KI-Tool
Genetische Veränderungen sind die treibende Kraft bei vielen Krebsarten, einschließlich der Leukämie. Sie haben einen starken Einfluss auf Prognose und Behandlung.
Ein neues KI-gestütztes Tool namens clinALL soll jetzt dazu beitragen, Ärzte bei der Diagnose und Behandlung pädiatrischer Leukämie zu unterstützen. Das Tool kombiniert genetische und klinische Daten in einer einfach zu bedienenden Plattform. Federführend beteiligt sind Professorin Dr. Anke K. Bergmann, leitende Oberärztin und stellvertretende Direktorin des Instituts für Humangenetik an der Medizinischen Hochschule Hannover (MHH) sowie Dr. Michelle Tang und Dr. Željko Antić. Das Team von Professorin Bergmann arbeitet kooperativ im internationalen Leibniz-Zukunftslabor für Künstliche Intelligenz (LeibnizAILab). Das Zukunftslabor wird vom Bundesministerium für Bildung und Forschung gefördert und von der Leibniz Universität koordiniert. Im Zukunftslabor arbeiten die Forschenden im Teilprojekt akute lymphatische Leukämie (ALL) daran, zusätzliche diagnostische und prognostische Marker für die ALL bei Kindern zu finden. Mit clinALL verfolgen sie einen innovativen Ansatz.
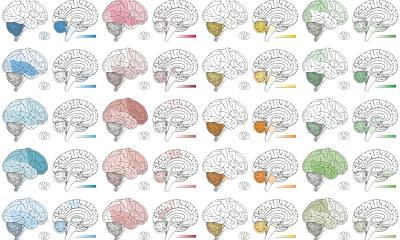
Es war uns auf diese Weise möglich, 78% der Patienten zu charakterisieren, die mit den gängigen Methoden nicht bestimmt werden konnten. [...] Somit haben wir insbesondere zur Klärung schwieriger Fälle beigetragen
Anke K. Bergmann
Die Studie umfasste 1.365 Patienten mit Blutkrebs, hauptsächlich pädiatrische Leukämie, bei denen eine genetische Routinediagnostik mit verschiedenen Standardtests durchgeführt wurde. Der zunehmende Umfang und die Komplexität von Sequenzierungsdaten zusammen mit anderen klinischen und diagnostischen Daten, wie zum Beispiel dem Ansprechen auf Medikamente oder die messbare Resterkrankung, stellen eine Herausforderung für eine effiziente klinische Erfassung und Interpretation dar. Das clinALL-Tool bietet ein durch Künstliche Intelligenz unterstütztes Gerüst, das genomische und klinische Daten in eine benutzerfreundliche Schnittstelle integriert. Auf diese Weise wird die Routinediagnostik unterstützt. Darüber hinaus können Erkenntnisse für bösartige Erkrankungen des Blut- und Lymphsystems gewonnen werden. Die Studienergebnisse finden somit direkt Anwendung in der klinischen Versorgung.
„Es war uns auf diese Weise möglich, 78% der Patienten zu charakterisieren, die mit den gängigen Methoden nicht bestimmt werden konnten. Auf der Abbildung sind diese Patienten grau markiert und den farblich markierten Subgruppen zugeordnet. Somit haben wir insbesondere zur Klärung schwieriger Fälle beigetragen“, sagt Professorin Bergmann, Humangenetikerin und Leiterin des Projektes „AI4ALL“ im Leibniz-Zukunftslabor mit Bezug auf das oben dargestellte Bild. Des Weiteren wurden mit clinALL auch andere wichtige Muster gefunden. So wurden zum Beispiel bestimmte Patientengruppen identifiziert, bei denen die Krankheit nach der Behandlung mit größerer Wahrscheinlichkeit zurückkehrt. Sowohl die Diagnostik als auch die Therapie werden präziser. Neue Fälle werden erkannt, und neue Subgruppen können identifiziert werden. Trotz der eher geringen Fallanzahl der Studie funktioniere das Tool sehr gut und liefere wichtige Erkenntnisse mit starkem Einfluss auf die Patientenversorgung. An der MHH ist clinALL bereits im Einsatz.
„Das Tool ermöglicht die Integration umfassender klinischer Daten mit erschwinglichen, gezielten Genttests. Somit ist clinALL auch für kleine Labore auch in Entwicklungsländern interessant“, betont Wissenschaftlerin Dr. Michelle Tang. Die Risikostratifizierung und Entscheidungsfindung vor Ort würden verbessert, und klinisch relevante Erkenntnisse würden zeitnah aufgezeigt werden. Das stellt einen bedeutenden Fortschritt in der personalisierten Medizin für Kinder mit Leukämie dar.
In der Studie wurde eine gezielte RNA-Sequenzierung von hämatologischen Neoplasien durchgeführt, in erster Linie bei pädiatrischer ALL in Kooperation mit der „AIEOP-BFM ALL“-Studiengruppe des Universitätsklinikums Schleswig-Holstein in Kiel, der „ALLTogether“-Studiengruppe des Universitätsklinikums Hamburg-Eppendorf und der pädiatrischen Hämatologie und Onkologie der MHH.
Quelle: Medizinische Hochschule Hannover; Text: Jana Illmer
29.07.2024