Historie
Dem Erreger der Justinianischen Pest auf der Spur
Aus einem bereits vor 50 Jahren in Süddeutschland geborgenen Skelett ist es nun erstmals gelungen, das Genom des Erregers der Justinianischen Pest vollständig zu rekonstruieren. Das Forschungsteam um Johannes Krause und Michal Feldman vom Max-Planck-Institut für Menschheitsgeschichte in Jena sowie Michaela Harbeck von der Staatssammlung für Anthropologie und Paläoanatomie in München, hat damit eine historische und hochqualitative Referenz geschaffen, die neue Einblicke in evolutionäre Schlüsselereignisse, Adaptionen und die Pathogenität eines Bakteriums erlaubt, das bis heute in vielen Teilen der Welt zu Pestausbrüchen führt.
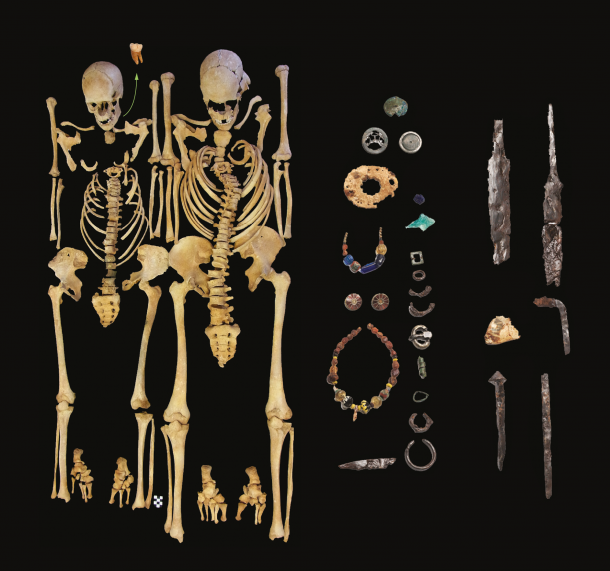

Die Justinianische Pest war die erste große historische Pest-Epidemie. Jahrhunderte vor dem berüchtigten „Schwarzen Tod“ tötete sie zur Zeit des Byzantinischen Reichs in der damals bekannten Welt nach Schätzungen bis zu 50 Millionen Menschen und trug möglicherweise zum Untergang des oströmischen Reiches bei. Bis heute sind die geographische Ausbreitung, die Mortalität und die Folgen der Justinianischen Pandemie nicht gänzlich geklärt. Neuere archäogenetische Studien haben jedoch gezeigt, dass für beide Pest-Pandemien der gleiche Erreger, das Bakterium Yersinia pestis, verantwortlich war. Das Rätsel, warum die Pest am Ende des 8. Jahrhundert zunächst wieder verschwand, bevor sie im 14. Jahrhundert als Schwarzer Tod zurückzukehrte, ist ungelöst. Einem Forschungsteam um Michal Feldman, Johannes Krause und Michaela Harbeck ist es nun erstmals gelungen die gesamte DNA des Bakteriums aus einem Skelett zu isolieren, das im 6. Jahrhundert unserer Zeitrechnung im Bayrischen Altenerding Opfer der Pest wurde. Das so gewonnene Genom datiert an den Anfang der Pandemie.
Altenerding – der erst zweite Fundort weltweit
Das jetzt untersuchte Skelett wurde aus einem so genannten Reihengräberfeld in Altenerding-Klettham nahe München geborgen und seit der Ausgrabung vor 50 Jahren in der Staatssammlung für Anthropologie und Paläoanatomie in München (SAPM) verwahrt. Vor der DNA-Analyse werden die Skelette dort morphologisch untersucht, dabei konnte der Anthropologe Bernd Trautmann feststellen, dass es sich bei dem Opfer um einen jungen Mann handelt, der gemeinsam mit einer jungen Frau in einem Grab bestattet wurde, weshalb die Vermutung nahe lag, dass sie Opfer einer schnell tötenden Infektionskrankheit wurden. Jetzt ist es Gewissheit, dass sie der Pest zum Opfer fielen. „Altenerding ist neben dem nur wenige Kilometer entfernten Gräberfeld Aschheim nun erst der weltweit zweite Fundplatz, auf dem der Erreger der Justinianischen Pest eindeutig nachgewiesen werden konnte“, sagt Andreas Rott, der zahlreiche Skelette aus Altenerding molekularbiologisch auf Pest prüfte, bevor er die DNA des Erregers gefunden hatte. „Wir freuen uns, dass wir nach der gelungenen Pest-Detektion in Aschheim, erneut die DNA von Yersinia pestis in unserem Sammlungsmaterial finden konnten“ sagt Michaela Harbeck, Konservatorin an der Staatssammlung für Anthropologie und Paläoanatomie. „Die damit ermöglichten Forschungsergebnisse machen die Wichtigkeit der fachgerechten Aufbewahrung archäologisch geborgener menschlicher Überreste für die Forschung deutlich“
Neue Einblicke in die Gefährlichkeit des Erregers
Der erneute Fund des Erregers in Süddeutschland bestätigt, dass die Justinianische Pest weit über die historisch überlieferten Regionen hinausreichte und erlaubt neue Einsichten in die Evolutions-geschichte von Yersinia pestis. „Damit wird das enorme Potential der Rekonstruktion von Genomen vergangener Pathogene demonstriert. Diese bieten nicht nur Einsichten in historische Prozesse, sondern vertiefen auch unser Verständnis der Erregerevolution“ sagt Michal Feldman, Genetikerin am Max-Planck-Institut für Menschheitsgeschichte in Jena und Erstautorin der Studie. Ein vor kurzem veröffentlichter Entwurf des Genoms der justinianischen Pest wird durch die neue Sequenz ergänzt und revidiert. „Wir konnten dreißig zusätzliche Mutationen und strukturelle Umlagerungen identifizieren, die einzigartig für den Justinianischen Erregerstamm sind“, erklärt Studienleiter Johannes Krause, Direktor am Jenaer Max-Planck-Institut. „Drei davon sind in Genen lokalisiert, die essentiell für die Virulenz, das heißt die krankheitserregende Kraft des Bakteriums sind.“ Außerdem zeigen die Daten, dass der Erregerstamm genetisch deutlich diverser war, als ursprünglich angenommen.
Die neuen Erkenntnisse erlauben es den Autoren, neue Richtlinien zu entwickeln, die dabei helfen könnten, die Qualität und Authentizität genomischer Daten, die von potentiellen früheren Krank-heitserregern gewonnen wurden, zu verbessern. Mit der Rekonstruktion des kompletten Genoms ist es zudem gelungen, eine wichtige historische und hochqualitative Referenz zu schaffen, mit deren Hilfe neue Einblicke in evolutionäre Schlüsselereignisse, Adaptionen und die Auswirkungen des Bakteriums auf den Menschen gewonnen werden können. Dies erscheint umso wichtiger, da die Pest in vielen Regionen der Welt als wiederkehrende Krankheit eingeordnet werden muss, die auch heute noch für zahlreiche Opfer sorgt.
Veröffentlichung
Michal Feldman, Michaela Harbeck, Marcel Keller, Maria A. Spyrou, Andreas Rott, Bernd Traut-mann, Holger C. Scholz, Bernd Päffgen, Joris Peters, Michael McCormick, Kirsten Bos, Alexander Herbig and Johannes Krause (2016) A high-coverage Yersinia pestis Genome from a 6th-century Justinianic Plague Victim, doi:10.1093/molbev/mswX
Quelle: Max-Planck-Institut für Menschheitsgeschichte
19.09.2016