© Tartila – stock.adobe.com
News • Vielversprechende Methode
Echtzeit-Genomik enttarnt Antibiotika-Resistenzen
Eine von Lara Urban, Principal Investigator bei Helmholtz AI und dem Pioneer Campus bei Helmholtz München und TUM Junior Fellow an der TUM School of Life Sciences, geleitete Studie hat eine neuartige Methode zur Erkennung versteckter Antibiotika-Resistenzen aufgezeigt, die neue Hoffnung auf wirksamere Behandlungen gegen resistente Infektionen bietet.
Veröffentlicht in Nature Communications, zeigt diese Forschung, wie Echtzeit-Genomik schnell und genau Antibiotikaresistenzen identifizieren kann, selbst in Fällen, in denen traditionelle Methoden versagen.
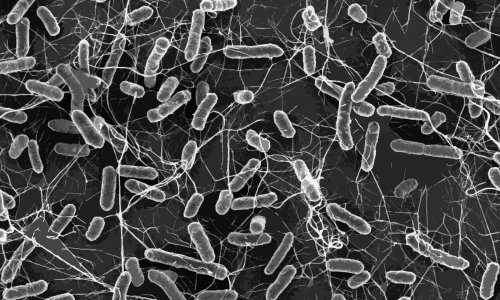
Antibiotika-Resistenz gehört zu den dringendsten Herausforderungen der modernen Gesundheitsversorgung und droht viele unserer wirksamsten Medikamente unbrauchbar zu machen. Diese globale Gesundheitskrise beeinträchtigt nicht nur die Patientenergebnisse, sondern belastet auch die Gesundheitssysteme mit höheren Kosten und längeren Krankenhausaufenthalten. Konventionelle Diagnostikmethoden basieren auf Kultivierung und phänotypischer Testung, die oft versagen, Resistenzen schnell und genau zu erkennen, insbesondere bei seltenen oder niedrigfrequenten Mechanismen. Die Verzögerungen bei der Kultivierung und anschließenden Testung können zu kritischen Verzögerungen bei der Verabreichung wirksamer Behandlungen führen, was sich erheblich auf die Genesungs- und Überlebensraten auswirkt.
Dieser Artikel könnte Sie auch interessieren

Artikel • AMR-Themenkanal
Antibiotikaresistenz: eine globale Bedrohung
Antibiotikaresistenz (AMR) stellt eine zunehmende Bedrohung für die öffentliche Gesundheit auf der ganzen Welt dar. Bakterien, die gegen verfügbare Antibiotika resistent werden, machen alltägliche medizinische Verfahren aufgrund des hohen Infektionsrisikos unmöglich. Lesen Sie weiter und erfahren Sie mehr über die AMR-Forschung, die Entwicklung neuer Antibiotika und Antibiotika-Alternativen.
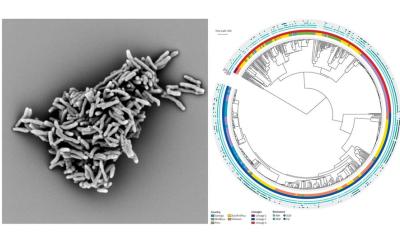
Die Studie zeigt bedeutende Fortschritte in der Echtzeit-Genomik durch Nanoporen-Sequenzierung, die eine schnellere und detailliertere Analyse von Pathogen-Genomen und Resistenzprofilen als herkömmliche diagnostische Werkzeuge bietet. Die Forschung konzentrierte sich auf eine schwere Infektion durch multiresistente Klebsiella pneumoniae, ein in Krankenhäusern häufig anzutreffendes Bakterium. Der Patient wurde zunächst basierend auf phänotypischen Suszeptibilitätstests mit Ceftazidim-Avibactam behandelt. Der Zustand des Patienten verschlechterte sich jedoch, da der Erreger Resistenzen entwickelte. Traditionelle Diagnostik konnte die genetischen Mechanismen dieser Resistenz nicht identifizieren.
Unsere Anwendung der Nanoporen-Sequenzierung zur Erkennung versteckter antimikrobieller Resistenzen unterstreicht nicht nur das Potenzial dieser Technologie zur schnellen Information der klinischen Versorgung weltweit, sondern zeigt auch, dass sie empfindlicher auf seltene Resistenzplasmide reagiert als etablierte Diagnostik
Lara Urban
Durch den Einsatz von Echtzeit-Genomik entdeckte das Team um Erstautorin Ela Sauerborn, PhD-Studentin bei Helmholtz Munich, komplexe Resistenzmechanismen, einschließlich einer neuartigen plasmid-kodierten Genvariante. Dieses Gen war vor der Behandlung in geringer Menge vorhanden und zeigt das Anpassungspotenzial des Pathogens unter Antibiotikadruck.
Die Integration von Echtzeit-Genomik in die klinische Praxis markiert einen bedeutenden Wandel im Management von Infektionskrankheiten. Schnelle, vor Ort durchgeführte Sequenzierungen ermöglichen es Gesundheitsdienstleistern, schnell maßgeschneiderte Behandlungsstrategien zu entwickeln, die auf das spezifische genetische Profil der Pathogene abgestimmt sind. Diese sofortige Einsicht in Resistenzprofile erlaubt es Klinikern, die wirksamsten Antibiotika zu verschreiben, Verzögerungen in der Behandlung zu minimieren und die Patientenergebnisse zu verbessern, während gleichzeitig die unnötige Exposition gegenüber Breitbandantibiotika reduziert wird.
Über die individuelle Versorgung hinaus verbessert die auf Echtzeit-Genomik basierende Resistenzerkennung die Infektionskontrolle und Eindämmungsstrategien. Die schnelle Bearbeitungszeit und die hohe Auflösung der Resistenzmechanismenerkennung ermöglichen gezielte und sofortige Eindämmungsmaßnahmen, die besonders in Gebieten mit hoher Resistenzbelastung entscheidend sind, um den Verlauf von Krankheitsausbrüchen zu ändern. „Unsere Anwendung der Nanoporen-Sequenzierung zur Erkennung versteckter antimikrobieller Resistenzen unterstreicht nicht nur das Potenzial dieser Technologie zur schnellen Information der klinischen Versorgung weltweit, sondern zeigt auch, dass sie empfindlicher auf seltene Resistenzplasmide reagiert als etablierte Diagnostik“, sagt Lara Urban.
Quelle: Helmholtz Munich
31.07.2024