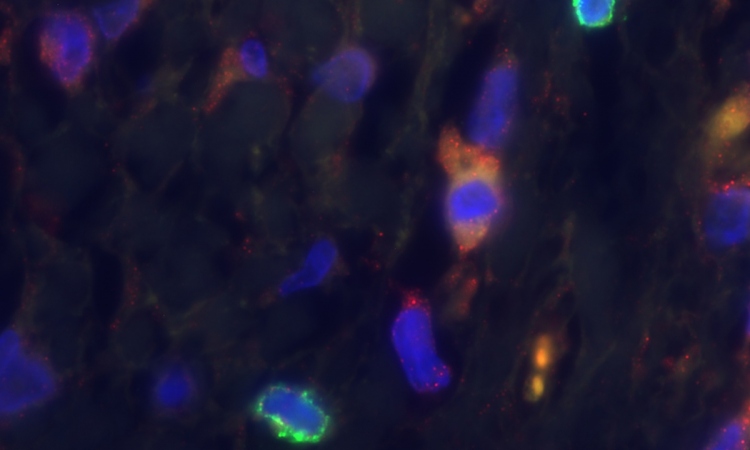
Spenderorgane
Lebern aus dem Labor ahmen natürliche Entwicklungsabläufe nach
Ein internationales Team von Wissenschaftlern des Max-Planck-Instituts für evolutionäre Anthropologie in Leipzig, des Cincinnati Children’s Hospital Medical Center, USA, und der Yokohama City University, Japan, hat herausgefunden, wie einzelne Zellen zusammenarbeiten und ihr Genom einsetzen, um sich in menschliches Lebergewebe zu entwickeln.
Die Studie ist ein Meilenstein auf dem Weg, mittels Biotechnologie gesundes und anwendbares menschliches Lebergewebe aus menschlichen pluripotenten Stammzellen herzustellen. Die Universität Leipzig stellte für die Auswertung der molekularbiologischen Daten umfangreiche Analyse-Werkzeuge bereit.
Die einzige heute mögliche Behandlung einer Lebererkrankung im Endstadium ist die Lebertransplantation. Doch die Anzahl der von verstorbenen Patienten gespendeten Lebern ist begrenzt. Ein Hauptziel der regenerativen Medizin ist es deshalb, selbstorganisierende menschliche Gewebe herzustellen, in denen Zellen eine Reihe koordinierter molekularer Ereignisse durchlaufen, die zeitlich präzise getaktet und räumlich präzise verortet sind, um funktionierende dreidimensionale Leberdivertikel herauszubilden. Dafür ist es unabdinglich, die während der Entwicklung des menschlichen Endoderms – aus dem sich später die Leber entwickelt – stattfindenden molekularen und zellulären Interaktionen bis ins kleinste Detail zu verstehen. „Transplantierbare Lebern und Lebergewebe biotechnologisch herzustellen, wäre ein großer Gewinn für Menschen, die an Lebererkrankungen leiden und innovative Behandlungen benötigen, um ihr Leben zu retten“, sagt Takanori Takebe, Forscher am Cincinnati Children’s Hospital Medical Center und der Yokohama City University. „Unsere Daten verschaffen uns jetzt ein neues, detailliertes Verständnis der interzellulären Kommunikation zwischen den sich entwickelnden Leberzellen und zeigen, dass wir menschliche Leberdivertikel herstellen können, die fetalen Zellen in ihrer natürlichen Entwicklung bemerkenswert ähnlich sind.“
In der aktuellen Studie nutzen die Forscher eine Technologie, die sich single-cell RNA sequencing (Einzelzellen RNA-sequenzierung) nennt und mit deren Hilfe sie messen können, wie individuelle Zellen sich verändern, wenn sie in einer dreidimensionalen Mikroumgebung miteinander kombiniert werden, in der Gefäßzellen, Bindegewebszellen und Leberzellen auf komplexe Art miteinander kommunizieren. „Der größte Vorteil der single-cell RNA sequencing-Technologie ist, dass sie uns für alle Zelltypen Genaktivitätskarten liefert, und wir so ihre Unterhaltung belauschen können“, sagt Keisuke Sekine von der Yokohama University, einer der beiden Erstautoren der Studie. Die Forscher konzentrierten sich darauf, für jede einzelne dieser Zellarten jeweils vor und nach ihrem Zusammentreffen zur Bildung von Leberkeimzellgewebe, einen kompletten Bauplan der aktiven Transkriptionsfaktoren, Signalmoleküle und -rezeptoren zu entwickeln. Dabei beobachteten sie, dass sich die Konversation und das Verhalten der Zellen drastisch verändern, sobald sie sich in der dreidimensionalen Umgebung zusammen entwickeln. „Es war aufregend, erstmals zu sehen, wie einzelne Zellen aufeinander reagieren, wenn sie in dieselbe Umgebung innerhalb der Leberkeimzelle gebracht werden. Das ähnelt dem Beobachten von Menschen auf einer Party“, sagt Gray Camp vom Max-Planck-Institut für evolutionäre Anthropologie, der andere der beiden Erstautoren der Studie.
Um das Verhalten der Zellen fundiert analysieren und interpretieren zu können, entwickelten Wissenschaftler vom Interdisziplinären Zentrum für Bioinformatik (IZBI) der Universität Leipzig ein umfangreiches Repertoire an Analyse-Werkzeugen und Softwareprogrammen. Kernstück der Auswertungen ist eine spezielle Methode maschinellen Lernens, sogenannte selbst-organisierende Karten (self-organizing maps, SOM). „Die SOM-basierten Analysen eignen sich gut für die Handhabung großer Datenmengen und für das Entflechten komplexer biologischer Zusammenhänge. Wir konnten die Methode bereits in umfangreichen Krebs-Patientenkohorten erfolgreich anwenden. Für das aktuelle Forschungsprojekt haben wir eine Vielzahl von Werkzeugen kombiniert. So konnten wir die Methode für die Single-Cell Analyse bestmöglich optimieren“, erklärt Henry Löffler-Wirth, Gruppenleiter am IZBI unter Leitung von Hans Binder. Das Softwareprogrammpaket wird nun erstmals mit der aktuellen Studie veröffentlicht. „Es bietet Forschergruppen weltweit freien Zugang zu den wichtigsten Werkzeugen für die Auswertung von Single-Cell Experimenten“, so Löffler-Wirth.
Die Single-cell RNA sequencing-Analyse half den Forschern auch dabei, die aus Stammzellen im Labor hergestellten dreidimensionalen Lebergewebe mit natürlich vorkommenden menschlichen fetalen und adulten Leberzellen zu vergleichen und ins Verhältnis zu bringen. Um diesen Vergleich zu ermöglichen, isolierte Georg Damm von der Arbeitsgruppe um Daniel Seehofer aus dem Bereich der translationalen Forschung der Leipziger Universitätsmedizin unter anderem primäre humane Hepatozyten aus Leberteilresektaten. Die Forscher beobachteten, dass ihre im Labor gezüchteten Leberdivertikel über molekulare und genetische Signaturen verfügten, die sehr stark den Signaturen ähneln, die in sich natürlich entwickelnden menschlichen Leberzellen vorhanden sind. „Unser Daten zeigen in einer vortrefflichen Auflösung, dass die Kommunikation zwischen Zellen unterschiedlichen Typs die einzelnen Zellen auf eine Art und Weise verändert, wie es möglicherweise auch während der natürlichen Entwicklung einer menschlichen Leber passiert“, sagt Barbara Treutlein. Insbesondere analysieren die Forscher die Wechselwirkung zwischen einem Signalprotein, das von Zellen produziert wird, um die Herausbildung von Blutgefäßen anzuregen (VEGF), und einem Rezeptorprotein, das mit VEGF kommuniziert, um die Blutversorgung innerhalb der sich entwickelnden Leber anzuregen (KDR). Wie die aktuelle Studie zeigt, liefert das Zusammenspiel dieser beiden Eiweiße äußerst wichtige Instruktionen zur Entwicklung und Reifung von Lebergewebe. Die Forscher schauten sich dieses Wechselspiel während der Entwicklung von Leberzellen bei Mäusen, natürlichen menschlichen Leberzellen und ihren biotechnologisch hergestellten Lebern an.
Dabei bemerkten die Autoren auch, dass die Genexpression – wo und wann genau Gene abgeschrieben werden – in den aus Stammzellen hergestellten Leberzellen nicht ganz mit der Genexpression bei natürlichen menschlichen Leberzellen übereinstimmt. Den verbleibenden Lücken zwischen natürlichen und biotechnologisch hergestellten Geweben liegen höchstwahrscheinlich Unterschiede in den Entwicklungssignalen zugrunde, die von der einzigartigen Mikroumgebung der Petrischale im Vergleich zum sich entwickelnden Menschen oder Tier ausgelöst werden. Die neuen zellulären und molekularen Daten der aktuellen Studie sollen zukünftig dazu dienen, die Leberknospen-Organoide weiterhin zu verbessern und die Differenzierung aller Zelltypen, die bei der Entwicklung der menschlichen Leber in Aktion treten, genauestens zu rekapitulieren. „Es gibt noch viel darüber zu lernen, wie man am besten eine funktionstüchtige menschlichen Leber in der Petrischale herstellt“, sagt Treutlein. „Doch unsere Studie ist ein großer Schritt in diese Richtung.“
Quelle: Universität Leipzig
20.06.2017