Video • Blick ins Hirn
Neuronale Netze bauen Karten des Gehirns
Der „Schaltplan“ eines Gehirns ist so komplex, dass Menschen ihn niemals ohne Hilfe nachbauen könnten. Jetzt haben Forscher einen Algorithmus geschrieben, der ihnen dabei hilft.
Um diesen Schaltplan, das sogenannte Konnektom eines Gehirns zu erstellen, erfassen Wissenschaftler von Google AI und dem Max-Planck-Institut (MPI) für Neurobiologie das Gehirn mit Hilfe von 3D-Elektronenmikroskopie-Aufnahmen. Mit einem Verfahren, das auf künstlichen neuronalen Netzen basiert, das ganze Nervenzellen mit allen Bestandteilen und Verbindungen nahezu fehlerfrei aus einem Bilderstapel herausarbeiten kann. Dieser Meilenstein in der automatischen Datenanalyse könnte die Kartierung ganzer Gehirne, und damit langfristig auch deren Verständnis, näherbringen.
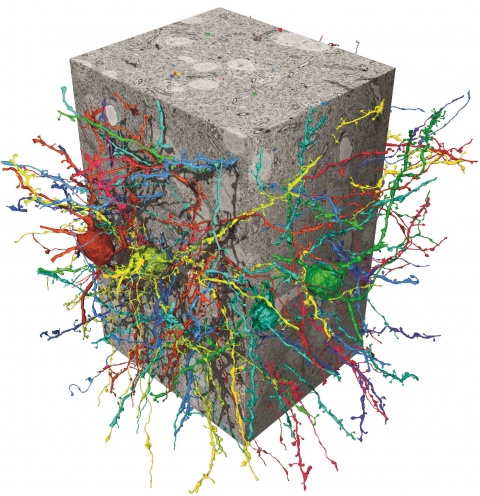
Im Vergleich zum Gehirn benutzen künstliche neuronale Netze stark vereinfachten "Nervenzellen". Dennoch hat die darauf basierende künstliche Intelligenz bereits unzählige Anwendungen gefunden: vom autonomen Fahren über die Qualitätskontrolle bis hin zur Diagnose von Krankheiten. Bei sehr komplexen Aufgaben, wie dem Herausarbeiten einzelner Nervenzellen mit all ihren Verästelungen und Kontaktstellen aus einer dreidimensionalen Bildaufnahme eines Gehirns, waren die Algorithmen bisher jedoch zu ungenau.
„Die Zellstrukturen, die der Computer aus unseren elektronenmikroskopischen Aufnahmen generierte, hatten einfach viel zu viele Fehler“, berichtet Jörgen Kornfeld vom Max-Planck-Institut für Neurobiologie in Martinsried. „Um damit etwas anfangen zu können, musste alles nochmals "korrekturgelesen" werden.“ Das nimmt viel menschliche Arbeitszeit in Anspruch: Ganze elf Jahre würde das Überprüfen eines Bilderstapels mit gerade mal 0,1 Millimetern Kantenlänge dauern. „Daher mussten wir etwas Besseres erfinden“, so Kornfeld. Besser als alles andere sind — zumindest derzeit – die flood-filling networks (FFNs), die Michal Januszewski zusammen mit seinen Kollegen bei Google A.I. entwickelt. Ein Datensatz aus dem Singvogelhirn, den Kornfeld schon vor Jahren aufgenommen und teilweise von Hand analysiert hatte, spielte bei dieser Entwicklung eine wichtige Rolle. Die wenigen, vom Menschen sorgfältig analysierten Zellen, stellen die Referenzdaten (ground truth) dar, anhand derer die FFNs erst lernten zu erkennen, wie ein richtiger Nervenzellausläufer aussieht, um mit dem Gelernten dann in Windeseile den Rest des Datensatzes zu kartieren.
Die Hochskalierung wird technisch sicher anspruchsvoll, aber im Prinzip haben wir jetzt im Kleinen demonstriert, dass alles Nötige für die Analyse bereitsteht
Jörgen Kornfeld
Die Zusammenarbeit zwischen Computerwissenschaftlern und Biologen hat in der von Winfried Denk geleiteten Abteilung schon Tradition. Der Leiter der Google Arbeitsgruppe, Viren Jain, war im Jahr 2005 Doktorand am MIT, als sich Denk an Jains Doktorvater, Sebastian Seung, wandte. Es ging um Hilfe bei der Analyse der mit einer in Denks Abteilung damals neu entwickelten Methode aufgenommenen Datensätze. In der Abteilung war Kornfeld damals damit beschäftigt, ein Computerprogramm für die Daten-Visualisierung und Annotation zu schreiben. Kornfeld, in dessen Forschung Neurobiologie und Datenwissenschaften zunehmend verschmelzen, entwickelte hauptverantwortlich das "SyConn" System zur automatischen Synapsenanalyse. Dieses wird – wie die FFNs – bei der Gewinnung biologischer Erkenntnisse aus dem Singvogeldatensatz unverzichtbar sein. Die Entwicklung der FFNs steht, so Denk, als Symbol für einen Wendepunkt in der Konnektomik. Die Geschwindigkeit der Datenanalyse hinkt nun nicht mehr der elektronenmikroskopischen Aufnahmegeschwindigkeit hinterher.
FFNs gehören zu den "Convolutional neural networks", einer speziellen Klasse von Algorithmen des automatischen Lernens. FFNs besitzen jedoch einen internen Rückkoppelungspfad, der es ihnen erlaubt auf bereits im Bild Erkanntes aufzubauen. Dies erleichtert es dem FFN enorm, die Unterscheidung von zellinternen und zellexternen Bereichen auf nahegelegene Bildelemente auszudehnen. Dabei lernt das FFN während der Lernphase nicht nur, welche Färbemuster eine Zellgrenze anzeigen, sondern auch, welche Formen diese Grenzen typischerweise haben. Die erwartete Einsparung an menschlicher Korrekturlesezeit durch die FFNs rechtfertigt auf jeden Fall, so Kornfeld, deren – im Vergleich zu bisherigen Methoden – größeren Verbrauch an Rechenleistung.
Inzwischen scheint es nicht mehr völlig undenkbar, wirklich große Datensätze, bis hin zu einem gesamten Maus- oder Vogelhirn, aufzunehmen und zu analysieren. „Die Hochskalierung wird technisch sicher anspruchsvoll, aber im Prinzip haben wir jetzt im Kleinen demonstriert“, sagt Jörgen Kornfeld, „dass alles Nötige für die Analyse bereitsteht.“
Quelle: Max-Planck-Institut für Neurobiologie
18.07.2018





