News • Dezentrale Plattform
"Codex"-Daten für bessere Covid-Forschung
Im Rahmen des Projekts "Covid-19 Data Exchange Platform (Codex)" stellt die Medizininformatik-Initiative (MII) des Bundesministeriums für Bildung und Forschung (BMBF) aktuelle Daten für die Covid-19-Forschung bereit. Das zeigt beispielhaft eine kürzlich veröffentlichte Studie der MII.
Die dezentrale Forschungsinfrastruktur der MII wird für Codex um eine zentrale Datenplattform erweitert, um der Covid-19-Forschung auf breiter Basis Real-World-Daten zur Verfügung zu stellen sowie Ärzten und Entscheidungsträgern im Gesundheitssystem tagesaktuell behandlungs- bzw. entscheidungsrelevante Daten zu Covid-19 zu liefern. Das im August 2020 gestartete Projekt ist zentraler Baustein des mit 150 Millionen Euro vom BMBF geförderten Netzwerks Universitätsmedizin (NUM).

In der MII haben seit 2018 bundesweit 29 Universitätskliniken sogenannte Datenintegrationszentren (DIZ) errichtet, um Daten aus der Versorgung und der Forschung klinikübergreifend und datenschutzgerecht für die Forschung nutzbar zu machen. Mit Beginn der Pandemie haben die DIZ frühzeitig begonnen, auch Covid-19-spezifische Daten strukturiert und in hoher Qualität für die Forschung bereitzustellen. Der Aufbau dieser dezentralen Infrastruktur hat einen schnellen Einstieg in die Entwicklung der Codex-Plattform im August 2020 ermöglicht.
Angestrebt wird, einen Covid-19-spezifischen Forschungsdatenbestand aller universitär-stationären Fälle zu erstellen. Diese standortübergreifende, auf internationalen Standards basierende Datengrundlage soll neuartige wissenschaftliche Auswertungen zu Covid-19 ermöglichen, zum Beispiel zum Einsatz und Erfolg von Therapien und Medikamenten oder zur Patientenstruktur an den Unikliniken. Dafür sollen möglichst kurzfristig pseudonymisierte klinische Daten, ergänzend auch Bilddaten und Daten zu Bioproben, aus allen deutschen Universitätskliniken zusammengeführt werden, wobei die erforderlichen ethischen und datenschutzrechtlichen Anforderungen berücksichtigt werden: Grundlage für die Datennutzung ist die Einwilligung der betreffenden Patientinnen und Patienten.
„Die umfassenden technischen und rechtlichen Vorarbeiten der Medizininformatik-Initiative, insbesondere die Datenintegrationszentren, sind wichtige Grundlage für die Forschungsdatenplattform zu Covid-19, obwohl wir uns erst mitten in der vom BMBF für 2018-2022 vorgesehenen Aufbauphase der MII befinden. Es ist wichtig, dass wir projektübergreifend öffentlich geförderte Forschungsinfrastrukturen gemeinsam nutzen und weiterentwickeln“, betont Sebastian C. Semler, TMF – Technologie- und Methodenplattform für die vernetzte medizinische Forschung e.V., Leiter der MII-Koordinationsstelle, der gemeinsam mit Ralf Heyder, Charité – Universitätsmedizin Berlin, Leiter der NUM-Koordinationsstelle, das Codex-Gesamtprojekt leitet.
Auf dieser Basis sind bereits erste wissenschaftliche Analysen zu Covid-19 publiziert worden, um den Herausforderungen der Pandemie zu begegnen. So demonstriert eine in dieser Woche veröffentlichte Studie des von der Friedrich-Alexander-Universität (FAU) Erlangen-Nürnberg geleiteten Forschungsteams der MII, wie Patientendaten der Uniklinikstandorte mit Hilfe der DIZ-Infrastruktur der MII mittels verteilter Auswertungskonzepte analysiert werden können. Untersucht wurde die Anzahl der stationären Krankenhauseinweisungen in 18 deutschen Universitätskliniken zwischen dem 1. Januar und 31. Mai 2020 im Vergleich zum selben Zeitraum 2018 und 2019.
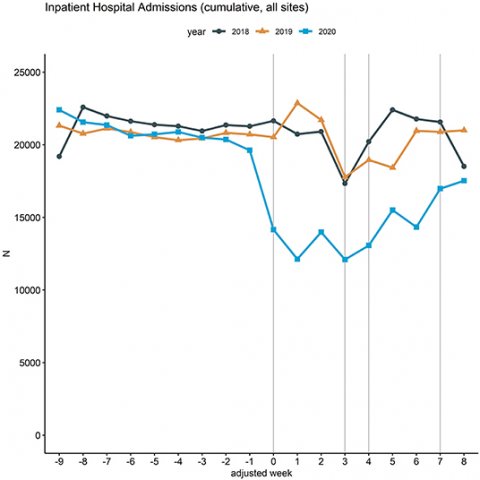
Bildquelle: Kapsner et al., Frontiers in Public Health 2021 (CC-BY 4.0)
Prof. Hans-Ulrich Prokosch, FAU Erlangen-Nürnberg, der die Entwicklung der dezentralen Komponenten der Codex-Plattform koordiniert, fasst die Studienergebnisse zusammen: „Die Anzahl der stationär aufgenommenen Patienten an den Universitätskliniken in Deutschland ist in den ersten vier Wochen nach der Ankündigung des Covid-19-Lockdowns am 16. März 2020 im Vergleich zu 2018 im Durchschnitt um 35 Prozent zurückgegangen. Das betrifft teilweise auch Krankenhauseinweisungen in Notfallaufnahmen, beispielsweise wegen eines Herzinfarkts. Hier wurde im Vergleich zum selben Zeitraum 2018 in 2020 eine um 38,7 Prozent reduzierte Anzahl von Patienten stationär aufgenommen. Der Unterschied war bei kritischen Gesundheitszuständen in der Krebsbehandlung deutlich geringer mit einer in 2020 nur um 13 Prozent reduzierten Krankenhausaufnahme im Vergleich zu 2018. Beim Vergleich zwischen Operationen zum Ersatz von Hüftgelenken bei Arthrose und zur Versorgung akuter Frakturen kann aber beispielhaft illustriert werden, dass planbare Operationen deutlich zurückgingen, wohingegen Notfalloperationen im Prinzip unverändert durchgeführt wurden.“
„Für Codex wird die erfolgreich aufgebaute dezentrale Forschungsdateninfrastruktur der MII um eine Plattform erweitert, die es Forschenden ermöglicht, komplexe Covid-19-Datensätze aus verschiedenen Quellen zentral auszuwerten: aus den Datenintegrationszentren der MII, aber auch aus kommunalen Kliniken, dem niedergelassenen Bereich und von Gesundheitsämtern. Die Plattform soll auch die Zusammenführung mit Daten ermöglichen, die direkt von Bürgerinnen und Bürgern über Apps erhoben werden“, erklärt Prof. Roland Eils, Zentrum für Digitale Gesundheit, Berlin Institute of Health (BIH) und Charité – Universitätsmedizin Berlin, der den Aufbau der zentralen Codex-Plattform koordiniert und wie Prof. Prokosch Mitglied des Codex-Lenkungsausschusses ist.
„Die Datenplattform soll insgesamt zu einem besseren Verständnis der Erkrankung Covid-19 beitragen und als Grundlage für politische Entscheidungen dienen. Außerdem wollen wir mit dieser Datenbasis die Entwicklung von neuen Diensten und Anwendungen für Gesundheitseinrichtungen und Bürger voranbringen und die Möglichkeit bieten, evidenzbasierte Entscheidungsunterstützungssysteme für Ärzte zu entwickeln“, erläutert Prof. Heyo K. Kroemer, Vorstandsvorsitzender der Charité und Mitinitiator des NUM.
Im Verlauf des Codex-Projekts, in dessen Startphase auch die klinische Forschungsplattform des Deutschen Zentrums für Herz-Kreislauf-Forschung (DZHK) zum Einsatz kommt, werden sukzessive weitere Funktionalitäten für die Covid-19-Pandemieforschung entwickelt und für den nachhaltigen Betrieb etabliert.
Quelle: TMF – Technologie- und Methodenplattform für die vernetzte medizinische Forschung
19.01.2021





