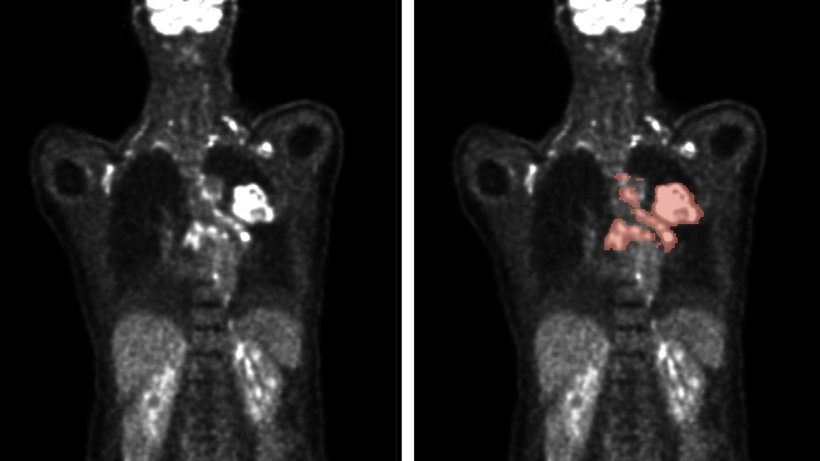
© Gatidis S, Kuestner T. (2022) A whole-body FDG-PET/CT dataset with manually annotated tumor lesions (FDG-PET-CT-Lesions) [Dataset]. The Cancer Imaging Archive. DOI: 10.7937/gkr0-xv29
News • Tumorerkennung in PET- und CT-Scans
Neue Ideen für die KI-gestütze Analyse medizinischer Bilder
Auf Deep Learning basierende Algorithmen können Tumoren erkennen – Forscher des KIT unter den besten Teams beim internationalen AutoPET Wettbewerb
Künstliche Intelligenz kann die Auswertung von medizinischen Bilddaten verbessern. So können auf Deep Learning basierende Algorithmen die Lage und Größe von Tumoren feststellen. Dies ist das Ergebnis von autoPET, eines internationalen Wettbewerbs zur medizinischen Bildanalyse. Forscher vom Karlsruher Institut für Technologie (KIT) erreichten den fünften Platz. Wie Algorithmen in der Positronen-Emissions-Tomographie (PET) und in der Computertomographie (CT) Tumorläsionen erkennen können, berichten die sieben besten autoPET Teams in der Zeitschrift Nature Machine Intelligence.
Bei der Diagnose von Krebs kommt bildgebenden Verfahren eine wesentliche Bedeutung zu. Die Lage, Größe und Art von Tumoren präzise zu bestimmen, ist entscheidend dafür, die passende Therapie zu finden. Zu den wichtigsten bildgebenden Verfahren gehören PET und CT. Erstere macht Stoffwechselvorgänge im Körper mithilfe von Radionukliden sichtbar. Bösartige Tumoren haben oft einen deutlich intensiveren Stoffwechsel als gutartige Gewebe. Verwendet wird dazu radioaktiv markierter Traubenzucker, meist Fluor-18-Desoxyglucose (FDG). Bei der CT wird der Körper in einer Röntgenröhre Schicht für Schicht durchleuchtet, um die Anatomie sichtbar zu machen und Tumoren zu lokalisieren.
Die Leistung [...] bei der Bilddatenauswertung hängt [...] auch vom Algorithmendesign [ab], beispielsweise was die Entscheidungen bei der Nachbearbeitung der vorhergesagten Segmentierung betrifft
Rainer Stiefelhagen
Krebspatienten haben teilweise Hunderte von Läsionen, das heißt durch das Wachstum von Tumoren verursachte krankhafte Veränderungen. Für ein einheitliches Bild gilt es, alle Läsionen zu erfassen. Mediziner bestimmen die Größe der Tumorläsionen, indem sie 2D-Schichtbilder manuell markieren – eine extrem aufwendige Arbeit. „Eine automatisierte Auswertung durch einen Algorithmus würde enorm Zeit sparen und die Ergebnisse verbessern“, erklärt Professor Rainer Stiefelhagen, Leiter des Computer Vision for Human-Computer Interaction Lab (cv:hci) des KIT.
Rainer Stiefelhagen und Zdravko Marinov, Doktorand am cv:hci, haben 2022 am internationalen Wettbewerb autoPET teilgenommen und unter 27 Teams mit 359 Teilnehmenden aus aller Welt den fünften Platz erreicht. Dabei bildeten die Karlsruher Forscher mit Professor Jens Kleesiek und Lars Heiliger vom IKIM – Institut für Künstliche Intelligenz in der Medizin in Essen ein Team. Organisiert vom Universitätsklinikum Tübingen und vom LMU Klinikum München, verband autoPET Bildgebung und maschinelles Lernen. Die Aufgabe bestand in der automatischen Segmentierung stoffwechselaktiver Tumorläsionen auf Ganzkörper-PET/CT. Für das Algorithmentraining hatten die teilnehmenden Teams Zugang zu einem großen kommentierten PET/CT-Datensatz. Alle für die letzte Phase des Wettbewerbs eingereichten Algorithmen basieren auf Methoden des Deep Learning. Dabei handelt es sich um einen Bereich des maschinellen Lernens, der vielschichtige künstliche neuronale Netze einsetzt, um komplexe Muster und Zusammenhänge in großen Datenmengen zu erkennen. Die sieben besten Teams aus dem Wettbewerb autoPET berichten nun in der Zeitschrift Nature Machine Intelligence über die Möglichkeiten automatisierter Auswertung medizinischer Bilddaten.
Wie die Forschenden in ihrer Publikation erklären, erwies sich ein Ensemble der bestbewerteten Algorithmen als überlegen gegenüber einzelnen Algorithmen. Das Algorithmenensemble kann die Tumorläsionen effizient und präzise erkennen. „Die Leistung der Algorithmen bei der Bilddatenauswertung hängt allerdings von der Quantität und der Qualität der Daten ab, aber auch vom Algorithmendesign, beispielsweise was die Entscheidungen bei der Nachbearbeitung der vorhergesagten Segmentierung betrifft“, erklärt Professor Rainer Stiefelhagen. Um die Algorithmen zu verbessern und robuster gegenüber äußeren Einflüssen zu machen, sodass sie sich im klinischen Alltag einsetzen lassen, sind weitere Forschungsarbeiten erforderlich. Ziel ist, die Analyse der medizinischen Bilddaten aus PET und CT in näherer Zukunft vollständig zu automatisieren.
Quelle: Karlsruher Institut für Technologie
22.11.2024