News • 4D-Bildgebung
Algorithmus rekonstruiert Prozesse aus Einzelbildern
Forscher am Helmholtz Zentrum München haben eine neue Methode entwickelt, um anhand von Bilddaten fortlaufende biologische Vorgänge, beispielsweise Krankheitsverläufe, zu rekonstruieren.
Die modernen Lebenswissenschaften liefern in immer kürzerer Zeit eine stets größer werdende Menge an Daten. Sie beherrsch- und auswertbar zu machen, ist das Ziel von Dr. Dr. Alexander Wolf und seinen Kollegen am Institute of Computational Biology (ICB) des Helmholtz Zentrums München. Dazu versuchen die Forscherinnen und Forscher, Software zu programmieren, die diese Auswertung übernimmt. Allerdings tun sich dabei verschiedene Hürden auf.
„In der aktuellen Studie haben wir uns mit der Fragestellung befasst, wie ein Algorithmus einzelne Bilder in einen kontinuierlichen Prozess einordnen kann“, erklärt Studienleiter Wolf. „So war es bisher zwar möglich, Bildinformation nach klar abgegrenzten Kategorien zu klassifizieren, bei Krankheitsverläufen oder in der Entwicklungsbiologie stößt das aber an Grenzen, weil die Prozesse keine Einzelschritte sondern eben fortlaufend sind.“
Um dem Rechnung zu tragen, bediente sich das Helmholtz-Team der Methode des sogenannten Deep Learning*, also maschinellen Lernprozessen. „Über künstliche neuronale Netze können wir nun Einzelbilder zu Prozessen zusammenrechnen und sie zudem für den Menschen nachvollziehbar abbilden“, sagen Philipp Eulenberg und Niklas Köhler, ehemalige Masterstudenten am ICB und Erstautoren der Studie.
Blutzellen und Netzhäute als Sparringspartner
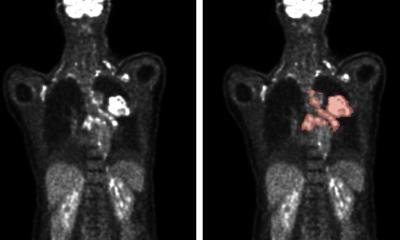
Um die Leistungsfähigkeit der Methode zu demonstrieren, wählten die Wissenschaftlerinnen und Wissenschaftler zwei Beispiele. Im ersten Ansatz rekonstruierte die Software den kontinuierlichen Zellzyklus von weißen Blutzellen anhand von Bildern aus einem bildgebenden Durchflusszytometer mit einem Fluoreszenzmikroskop. „Ein weiterer Vorteil dieser Betrachtung liegt darin, dass unsere Software so schnell ist, dass man die Entwicklung der Zellen quasi 'on-the-fly' also noch während der Analyse im Zytometer abbilden kann“, erklärt Wolf. „Darüber hinaus macht unsere Software sechsmal weniger Fehler als bisherige Ansätze.“
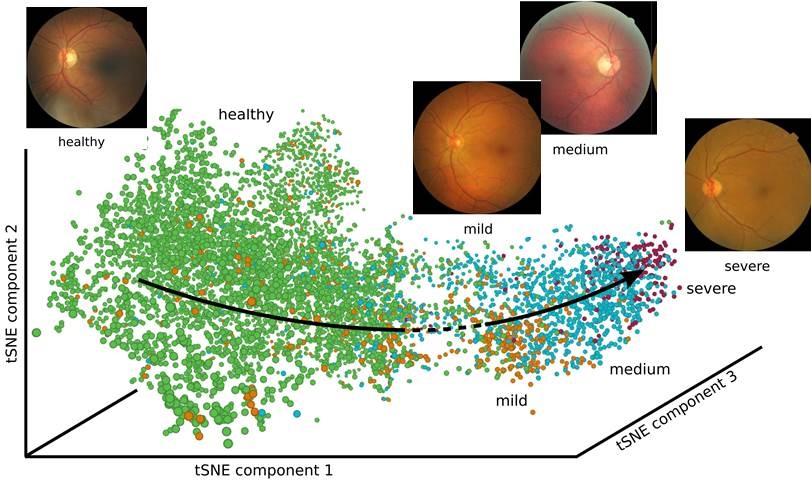
Im zweiten Experiment rekonstruierten die Forscher den Krankheitsverlauf einer diabetischen Retinopathie.** „Dazu gaben wir unserer Software 30.000 einzelne Bilder von Netzhäuten - sozusagen als Sparringspartner“, erklärt Niklas Köhler. „Dadurch, dass sie die Daten automatisch zu einem kontinuierlichen Prozess zusammenfügt, erlaubt uns die Software, eine Vorhersage für den Krankheitsverlauf auf einer kontinuierlichen Skala zu treffen.“
Und sollten die Daten nicht in einen fortlaufenden biologischen Prozess gehören? „In einem solchen Fall erkennt die Software, dass es sich um ungeordnete Einzelkategorien handelt und verteilt die Messdaten auf einzelne Cluster“, so Wolf. Neben weiteren Anwendungen der Methode wollen Wolf und seine Kollegen in Zukunft weitere Probleme bei der Auswertung biologischer Daten mit Hilfe von maschinellem Lernen lösen.
Quelle: Helmholtz Zentrum München - Deutsches Forschungszentrum für Gesundheit und Umwelt
* Algorithmen des Deep Learning simulieren Lernprozesse, wie sie beim Menschen vorkommen (neuronale Netze) – in etwa so wie ein Kind lernt, Gesichter zu erkennen oder Tiere zu unterscheiden. Das Prinzip funktioniert besonders gut, wenn große Datenmengen (Big Data) zum Training verfügbar sind. Eine der Stärken von Deep Learning ist die Bilderkennung. Zwischen der Eingabe und der Ausgabe sind hier mehr Entscheidungsebenen (layers) zwischengeschaltet als sonst bei neuronalen Netzen üblich, daher der Begriff der Tiefe.
** Diabetische Retinopathie ist die Hauptursache für den frühen Verlust des Augenlichts in der westlichen Welt. Die Diagnose erfolgt normalerweise durch Fachpersonal, was die vier Stadien gesund, mild, mittel und schwer zuordnet. Die Software konnte anhand von 8000 Bildern den Verlauf beziehungsweise die zunehmende Schwere der Krankheit beschreiben, ohne dass sie Informationen zur Ordnungsfolge bekommen hatte.
09.09.2017