Bildquelle: Goethe-Universität Frankfurt
News • Künftige Coronavirus-Generationen
So bereiten sich Forscher auf “SARS-CoV3” vor
Ein internationaler Forschungsverbund hat erstmals RNA-Faltungsstrukturen des SARS-CoV2-Genoms beobachtet, mit denen das Virus den Infektionsverlauf steuert.
Da diese Strukturen bei verschiedenen Beta-Coronaviren sehr ähnlich sind, legen die Wissenschaftler damit nicht nur die Grundlagen für die gezielte Entwicklung neuartiger Medikamente zur Covid-19-Behandlung, sondern auch für Infektionsgeschehen mit neuen Coronaviren, die sich in der Zukunft entwickeln könnten.
Die Forscher veröffentlichten ihre Ergebnisse jetzt im Fachjournal Nucleic Acids Research.
Wir hoffen, dass wir einen Beitrag dazu leisten konnten, auf künftige ‚SARS-CoV3‘-Viren besser vorbereitet zu sein
Harald Schwalbe
Genau 29.903 Buchstaben lang ist der genetische Code des SARS-CoV2-Virus, aufgereiht in einem langen RNA-Molekül. Es beinhaltet die Information zur Herstellung von 27 Proteinen. Dies ist zwar sehr wenig im Vergleich zu den vielleicht 40.000 Proteinsorten, die eine menschliche Zelle herstellen kann. Doch Viren nutzen bekanntermaßen ja den Stoffwechsel ihrer Wirtszellen, um sich selbst zu vermehren. Entscheidend ist für diese Strategie, dass Viren die Bildung der eigenen Proteine präzise steuern können.
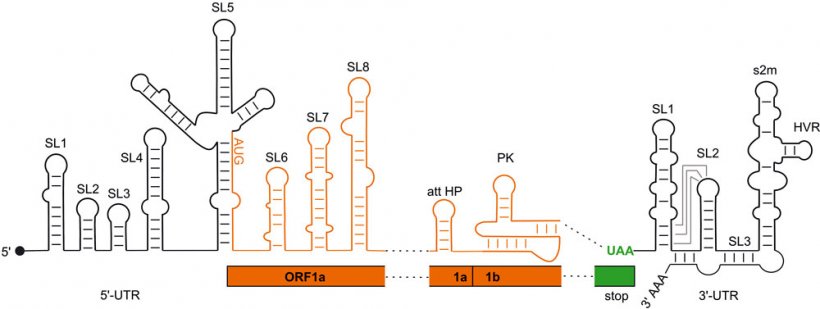
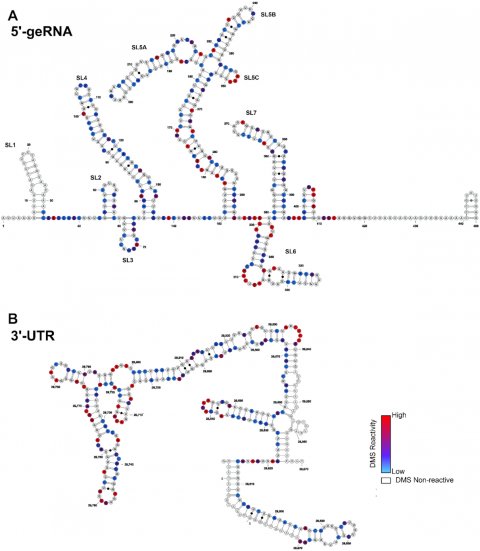
SARS-CoV2 nutzt als Steuerelemente für die Protein-Herstellung räumliche Faltungen seines RNA-Erbmoleküls: Überwiegend in Bereichen, die nicht für die Viren-Proteine codieren, werden aus dem RNA-Einzelstrang Strukturen mit RNA-Doppelstrang-Abschnitten und –schleifen. Bisher gab es allerdings nur Modelle dieser Faltungen, die sich auf Computeranalysen oder indirekte experimentelle Nachweise stützten.
Nun konnte ein internationales Team von Wissenschaftlern unter Leitung von Chemikern und Biochemikern der Goethe-Universität und der TU Darmstadt die Modelle erstmals experimentell überprüfen. Beteiligt waren außerdem Forscher des israelischen Weizmann Institute of Science, des schwedischen Karolinska-Instituts und der Katholischen Universität Valencia.
Bildquelle: Wacker et al., Nucleic Acids Research 2020 (CC BY-NC 4.0)
Die Forscher konnten die Struktur von insgesamt 15 solcher regulatorischen Elemente bestimmen. Dazu nutzten sie die Kernresonanz- oder NMR-Spektroskopie, bei der die Atome der RNA einem starken Magnetfeld ausgesetzt werden und so etwas über ihre räumliche Anordnung verraten. Die Ergebnisse dieser Methode glichen sie mit denen aus einem chemischen Verfahren ab (Dimethylsulfat-Footprint), mit dessen Hilfe zwischen RNA-Einzelstrang- und Doppelstrangbereichen unterschieden werden kann.
Der Koordinator des Konsortiums, Prof. Harald Schwalbe vom Zentrum für Biomolekulare Magnetische Resonanz der Goethe-Universität Frankfurt, erläutert: „Mit unseren Ergebnissen haben wir eine breite Basis gelegt, um künftig genau zu verstehen, wie SARS-CoV2 das Infektionsgeschehen steuert. Wissenschaftlich war das ein gewaltiger, sehr arbeitsintensiver Kraftakt, den wir nur durch den außergewöhnlichen Einsatz der Teams hier in Frankfurt und Darmstadt gemeinsam mit unseren Partnern im Covid-19-NMR-Konsortium stemmen konnten. Doch es geht direkt weiter: Derzeit untersuchen wir zusammen mit unseren Kooperationspartnern, welche viralen Proteine und welche Proteine der menschlichen Wirtszellen mit den gefalteten regulatorischen Regionen der RNA interagieren, und ob sich daraus Ansatzpunkte für Therapien ergeben können.“
Weltweit forschen über 40 Arbeitsgruppen mit 200 Wissenschaftlern im Covid-19-NMR-Konsortium, in Frankfurt arbeiteten seit Ende März 2020 45 Doktoranden und Postdocs in zwei Schichten pro Tag an sieben Tagen die Woche mit.
Das Potenzial der Entdeckungen geht über neue Behandlungsoptionen für Infektionen mit SARS-CoV2 hinaus, davon ist Schwalbe überzeugt: „Die Steuerungsregionen der viralen RNA, deren Struktur wir untersucht haben, sind zum Beispiel bei SARS-CoV fast identisch und auch bei anderen Beta-Coronaviren sehr ähnlich. Daher hoffen wir, dass wir einen Beitrag dazu leisten konnten, auf künftige ‚SARS-CoV3‘-Viren besser vorbereitet zu sein.“
Quelle: Goethe-Universität Frankfurt
23.11.2020