Quelle: Scientific Reports
News • Künstliche Intelligenz
Machine Learning steigert biotechnologische Proteinproduktion
Forscherinnen und Forscher des Paul-Ehrlich-Instituts (PEI) haben in einer Forschungskooperation ein mathematisches Modell entwickelt, mit dem eine präzisere Vorhersage und eine verbesserte Ausbeute bei der biotechnologischen Proteinsynthese in Wirtsorganismen möglich ist. Das neue Verfahren bietet vielfältige biotechnologische Anwendungsmöglichkeiten u.a. in der Impfstoffentwicklung.
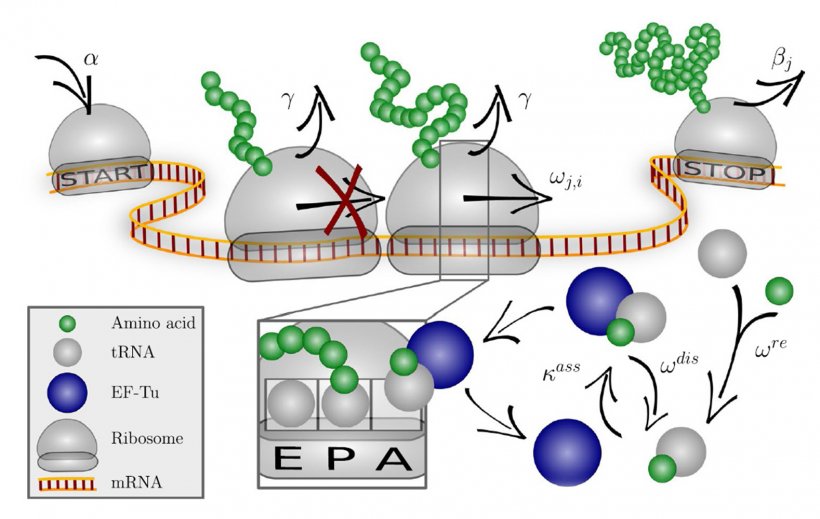
Biotechnologische Arzneimittel beruhen häufig auf maßgeschneiderten Proteinen, die in Zellkulturen oder in Bakterien produziert werden. Hierzu werden die Gene mit den Informationen über die Aminosäuresequenz der gewünschten Proteine in Bakterien- oder Säugerzellen transferiert. Oft reicht dies aber nicht aus, damit im gewünschten Umfang die transferierten Gene abgelesen und die darauf kodierten Proteine gebildet werden. Meist ist zusätzlich eine Anpassung der betreffenden Gene an die Wirtszelle erforderlich. Dies passiert u.a. durch die Anpassung des Codes für die Aminosäuren. Die Reihenfolge von je drei Nukleobasen der Boten-RNA (mRNA), auch Codon genannt, legt die einzelnen Aminosäuren fest, die Abfolge der Codons legt die Aminosäuresequenz der Proteine fest. Ein Austausch dieser Codons ist deshalb erforderlich, weil unterschiedliche Organismen bzw. Zellsysteme unterschiedliche Codons für ein- und dieselbe Aminosäure präferieren. Warum, ist wissenschaftlich nicht vollständig geklärt. Die Anpassung der Codons erfolgt daher bisher nach einem heuristischen Ansatz.
Wie lässt sich besser vorhersagen, welche Optimierungsschritte geeignet sind? Wissenschaftlerinnen und Wissenschaftler um Dr. Jan-Hendrik Trösemeier und Dr. Christel Kamp, Fachgebiet Biostatistik der Abteilung Mikrobiologie des Paul-Ehrlich-Instituts, untersuchten in einer von der Adolf-Messer-Stiftung unterstützten Forschungskooperation mit Forschenden des Max-Planck-Instituts für Kolloid- und Grenzflächenforschung, Potsdam, und der Goethe-Universität Frankfurt/Main die Proteinexpression im sogenannten Codon-spezifischen Elongationsmodell (COSEM). Darin wird mit mathematischen Methoden die Dynamik der Synthese der Proteine (Proteintranslation) in den entsprechenden Zellen simuliert und eine Codon-spezifische Proteinsyntheserate abgeleitet.
Mit den Daten dieser Simulation haben die Forschenden unter Berücksichtigung weiterer Prädiktoren für die Protein-Ausbeute mit Methoden des maschinellen Lernens („machine learning“) den sogenannten Proteinexpressions-Score ermittelt. Dieser dient zur Vorhersage der Proteinausbeute und zur Codon-Optimierung der Gene, die in fremden Zellen (heterolog) exprimiert werden. In verschiedenen Modellorganismen wiesen die Forscherinnen und Forscher nach, dass ihre simulationsunterstützte Optimierungsmethode herkömmlichen Verfahren überlegen ist. Mit dem neu entwickelten, modular aufgebauten Modell lässt sich nicht nur die Proteinausbeute steigern, sondern es können weitere Optimierungen erfolgen, zum Beispiel die Verbesserung der Translationsgenauigkeit.
Der Algorithmus ist in einer speziellen Software implementiert und erlaubt die oben beschriebene benutzerdefinierte Optimierung von Genen. Der Algorithmus kann aber auch für den gegensätzlichen Weg genutzt werden, die Deoptimierung. Wozu dient sie? Eine solche Deoptimierung von Genen kann beispielsweise genutzt werden, um Pathogene genetisch zu verändern und abzuschwächen. Diese Abschwächung – Attenuierung – von Pathogenen wird bei der Entwicklung von Impfstoffen genutzt: Lebendimpfstoffe werden vom ursprünglichen Pathogen abgeleitet und sind genetisch so verändert, dass sie im Menschen zwar eine Immunreaktion erzeugen, sich aber nur noch begrenzt vermehren und keine Krankheit mehr auslösen können.
Quelle: Paul-Ehrlich-Institut - Bundesinstitut für Impfstoffe und biomedizinische Arzneimittel
20.05.2019