Artikel • Oberflächenverstärkte Raman-Spektroskopie
Lab-on-a-Chip-Ansätze in der medizinischen Diagnostik
Der analytisch sensitive und spezifische Nachweis von Medikamenten oder von Metaboliten in Körperflüssigkeiten sowie der schnelle und sichere Nachweis von humanpathogenen Krankheitserregern stellt die instrumentelle Analytik im Bereich der medizinischen Diagnostik vor große Herausforderungen.
Institutionsangaben der Authoren
1. Leibniz-Institut für Photonische Technologien (IPHT); 2. Friedrich Schiller Universität Jena, Institute of Physical Chemistry and Abbe Centre of Photonics; 3. InfectoGnostics Science Campus; 4. Universitätsklinikum Jena (UKJ); 5. Klinikum rechts der Isar der TU München, Institut für klinische Chemie und Pathobiochemie
In den letzten Jahren hat sich herauskristallisiert, dass die Kombination der oberflächenverstärkten Raman-Spektroskopie (surface-enhanced Raman spectroscopy, SERS) mit mikrofluidischen Funktionselementen (Lab-on-a-Chip) hervorragend geeignet ist, automatisierte und reproduzierbare Messbedingungen zu gewährleisten und/oder kleine Probenvolumina im Nanoliter-Bereich zu analysieren.1
Die SERS-Technologie ist gekennzeichnet durch den molekular-spezifischen Nachweis kleinster Konzentrationen pharmazeutischer Substanzen. Dabei wird die erforderliche Sensitivität durch die Verwendung plasmonisch-aktiver metallischer Nanostrukturen wie z.B. kolloidale Silber- und Goldnanopartikel oder nano- und mikrostrukturierte Metalloberflächen erreicht. Zugleich kann diese Methode direkt, d.h. ohne die Anwendung aufwendiger Aufreinigungsprotokolle, für den Nachweis einzelner pharmazeutischer Wirkstoffe oder Metabolite in komplexen humanen Matrices wie Urin, Blutplasma oder Sputum im Konzentrationsbereich von wenigen mg/mL bis unter µg/mL eingesetzt werden.
Ungeachtet des herausragenden Potentials der SERS-Methode für bioanalytische Fragestellungen ist diese in der medizinischen Routinediagnostik heute noch nicht etabliert. Dies ist vor allem auf die hohen Qualitätsanforderungen an die SERS-aktiven Nanostrukturen zurückzuführen, die eine reproduzierbare Signalintensität gewährleisten müssen. Durch die Verwendung von Mikrofluidik- bzw. Lab-on-a-Chip-Ansätzen können die Limitierungen der SERS-Methode wie unkontrollierte Aggregation der kolloidalen Metallnanopartikel überwunden werden.
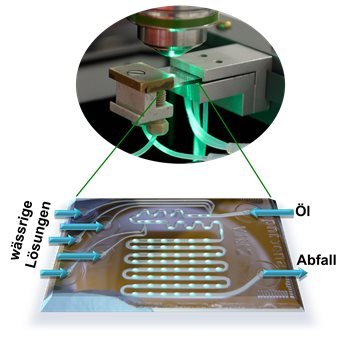
Die zur Verfügung stehenden Lab-on-a-Chip-SERS-Plattformen können in zwei Kategorien eingeteilt werden: Durchfluss oder kontinuierliche Durchfluss-Plattformen sowie (ii) segmentierte bzw. Tropfen-basierte Ansätze.1 Im Falle der Durchfluss-Plattformen werden zum Beispiel metallische Kolloidnanopartikel mit den zu analysierenden Proben vermischt oder feste (stationäre), metallische Nanostrukturen in das Kanalsystem integriert. Im Vergleich zu herkömmlichen SERS-Messungen auf offenen Plattformen wird die Flüssigkeitsverdampfung vermieden. Nachteilig ist, dass die Kanalwände permanent mit der Probenflüssigkeit in Kontakt stehen und somit ein sog. Memory-Effekt* auftreten kann.
*Als Memory-Effekt wird die Anreicherung von Analytmolekülen an den Kanalwänden im Laufe der Messzeit bezeichnet, was zur Überbestimmung der Konzentration führen kann.
Durch die Verwendung eines segmentierten Flusses, vor allem bei Tropfen-basierten SERS-Ansätzen (dargestellt in Abbildung 1), kann diese Kreuzkontamination verhindert werden, da zum Beispiel wässrige Tropfen in einer Ölmatrix transportiert werden und ein Kontakt der Probenlösung und der metallischen Kolloide mit der Kanalwand verhindert wird. Jeder Tropfen kann als individuelle Messzelle oder Mikroreaktor angesehen werden. Es kann somit erreicht werden, dass automatisiert eine große Anzahl von Spektren in einer kurzen Zeit zur Darstellung einer umfangreichen Datenbank aufgenommen werden.
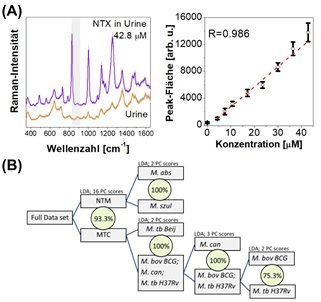
Wir konnten in unserer Arbeitsgruppe die Leistungsfähigkeit der Lab-on-a-Chip-SERS-Technologie unter Verwendung eines Tropfen-basierten Ansatzes für den Nachweis von Medikamenten und Metaboliten in Körperflüssigkeiten sowie für die Identifikation von Mykobakterien unter Beweis stellen. Dazu wurden humane Urinproben mit Levofloxacin, einem Antibiotikum, versetzt und Konzentrationen im Bereich zwischen 0,45 mM (162,6 μg/mL) und 1,8 mM (650,5 μg/mL) erfolgreich nachgewiesen.2 Laut Literatur ist die erwartete Levofloxacinkonzentration im Urin 1,38 mM ± 0,68 mM mit einer gemessenen Mindestkonzentration von 0,45 mM nach 4 h nach der Verabreichung einer 500 mg Dosis. Weiterhin konnte am Beispiel des Nachweises von Nitroxolin (NTX) in humanen Urinproben der Einsatz der Standardadditionsmethode erfolgreich gezeigt werden.3 In Abbildung 2A ist ein typisches SERS-Spektrum von NTX sowie die konzentrationsabhängige Evolution der Raman-Intensität einer Markerbande dargestellt. Die vorgestellten Ergebnisse zeigen daher, dass Lab-on-a-Chip-SERS ein vielversprechendes bioanalytisches Werkzeug zur Urinanalyse ist.
Weiterhin konnte gezeigt werden, dass die Enzymaktivität von Thiopurin-Methyltransferase (TPMT) in lysierten roten Blutkörperchen bestimmt werden kann.4 Hier bei wird spektroskopisch der Umsatz des Medikaments 6-Mercaptopurin zu 6-Methylmercaptopurin verfolgt und Rückschlüsse auf die TPMT-Aktivität gezogen. Mit einer Genauigkeit von 96% kann zwischen keiner und normaler TPMT-Aktivität unterschieden werden.
Im Rahmen des Nachweises von bakteriellen Krankheitserregern konnte gezeigt werden, dass mit Hilfe von SERS der für Pseudomonas aeruginosa spezifische Metabolit Pyocyanin quantifiziert werden kann.5 Unter Verwendung eines robusten SERS-Substrates konnte Pyocyanin in artifiziellem Sputum abhängig von der Konzentration im relevanten Bereich bei Mukoviszidose-Patienten, die besonders häufig unter diesem pathogenen Keim leiden, nachgewiesen werden.6
Schließlich wurde auch der Lab-on-a-Chip-SERS-Ansatz für die Identifikation von Mykobakterien eingesetzt, um dem Arzt eine Entscheidungshilfe für die zu verwendenden Medikamente zur Verfügung zu stellen.7 In Abbildung 2B ist das chemometrische Modell zur bakteriellen Identifikation dargestellt.
Somit kann festgehalten werden, dass die Lab-on-a-Chip-SERS-Technologie ein hohes Potential für medizinische Anwendungen aufweist. In zukünftigen Arbeiten soll dies durch die Verwendung von Realproben unter Beweis gestellt werden.
Quellen:
27.09.2019