Interview • Zwischen Mensch und Molekül
Risikogene per Klick entschlüsseln
An der Schnittstelle zwischen Mensch und Molekül forscht Prof. Dr.-Ing. Christoph M. Friedrich beinahe täglich.
Interview: Daniela Zimmermann
Der gebürtige Westfale ist Professor für biomedizinische Informatik an der Fachhochschule Dortmund und seit kurzem auch am Institut für Medizinische Informatik, Biometrie und Epidemiologie (IMIBE) am Universitätsklinikum Essen tätig. Die Kooperation der Einrichtungen hat bereits 2013 einen gemeinsamen Masterstudiengang der Fachhochschule und der Universität Duisburg-Essen hervorgebracht. Welche Vorteile die Arbeit am Institut mit sich bringt, wann die medizinische Bioinformatik ins Spiel kommt und wo die Schnittstelle zur Radiologie liegt, verrät Friedrich im Interview mit European Hospital.
Womit beschäftigt sich die medizinische Bioinformatik?

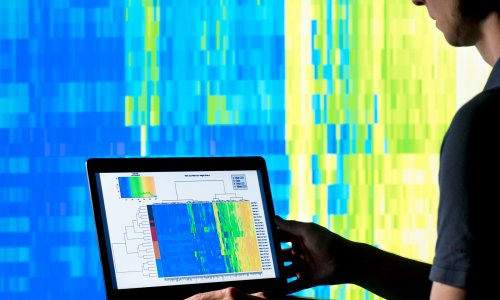
Die medizinische Bioinformatik beschäftigt sich nicht nur mit der Datensammlung, -aufbereitung und -verwertung im medizinischen Alltag, sondern verknüpft die Analyse von biologischen Präparaten und Proben wie zum Beispiel die Gen-Analyse mit intelligenten Auswertungs- und Interpretationssystemen. Dabei spielt auch die Bildgebung eine wichtige Rolle. Aktuell forschen wir zum Thema automatische Knochenaltersbestimmung anhand von Röntgenbildern; weitere Forschungsarbeiten auf der Grundlage von MRT- und CT-Aufnahmen stehen auf der Agenda.
Sie greifen also dem Radiologen unter die Arme?
Generell ist die Forschung in diesem Bereich in zweierlei Hinsicht unterstützend. Viele radiologische Arbeiten sind mühselig, Quantität und Aufwand steigen immer weiter an, wodurch sich natürlich auch die Chance erhöht, etwas zu übersehen. Die KI-gestützte Diagnose kann dem Radiologen wichtige Hinweise liefern und im Zweifelsfall die Fehlerrate senken. Der zweite Bereich ist die Arbeitsbeschleunigung. Gerade Schichtaufnahmen fressen Zeit, bis der Radiologe zum interessanten Punkt kommt. Mithilfe von Software und anatomischen Informationen kann ich ihn direkt zu dieser Stelle leiten. Hier kommt die strukturierte Befundung zum Tragen. Natürlich sollte stets der Facharzt die finale Anpassung eines Befunds übernehmen, aber dank automatischer Analyse könnten bestimmte Formularfelder bereits vorausgefüllt werden und ihm Arbeit ersparen.
Ideen wie die strukturierte Befundung via KI sind nicht neu...
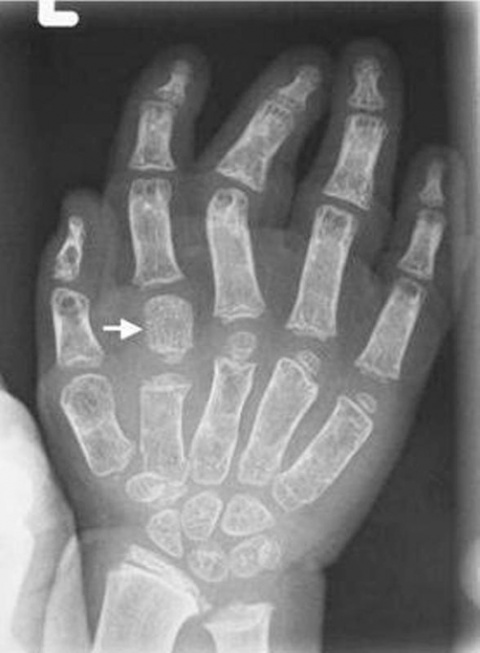
Nein, aber die Forschungsansätze und Methoden haben sich verändert. Bis vor fünf oder sechs Jahren hat das maschinelle Lernen mit einem klassischen, feature-basierten Ansatz stattgefunden. Das heißt, man versuchte aus radiologischen Aufnahmen bestimmte Features wie beispielweise Histogramme der Grauwertverteilung zu extrahieren und auszuwerten. Das war vergleichsweise erfolgreich und viele kommerzielle Produkte arbeiten auch heute noch auf dieser Basis. Vor einigen Jahren begann jedoch der Deep Learning Hype und hat die bisherigen Ansätze in Sekundenschnelle überholt. Convolutional neural networks, also Faltungsnetzwerke für Vorhersagen zu nutzen, macht einen großen Unterschied in Bezug auf Einfachheit, Schnelligkeit und Zuverlässigkeit von Studienergebnissen. Plötzlich erhielt man in kürzester Zeit Daten, für die man zuvor monatelang Herangehensweisen testen, anpassen und validieren musste. Der Anfang einer Reise, die noch lange nicht am Ende ist. Denn wir stellen verstärkt fest, dass die Zukunft nicht im reinen Deep Learning liegt, sondern in einer geschickten Methodenkombination aus Deep Learning und Text-Mining.
Welche Vorteile bietet diese Kombination?
Koppelt man Bild und textuelle Beschreibung, führt das auf jeden Fall zu einer besseren Klassifikationsrate und spezifischeren Vorhersagen. Wir haben mit einem ähnlichen Forschungsansatz bereits gute Ergebnisse erzielt. Dabei haben wir anhand von frei erhältlichen, radiologischen Abbildungen aus medizinischen Publikationen mit zugehörigen Bildunterschriften ein Faltungsnetzwerk trainiert, das im Anschluss präzise Bildunterschriften für die entsprechenden Bilder vorhersagte. Das Programm konnte Aussagen treffen wie „in dieser speziellen anatomischen Region ist eine Fraktur zu sehen“ und weitere Schlüsselwörter dazu angeben.
Eine Software auf dieser Basis könnte also bei der Befundung unterstützen. Hierbei spielt aber auch die textuelle Geschichte des Patienten eine Rolle. Müssten deshalb nicht weitere Informationen wie Alter, Geschlecht, Cholesterinwerte etc. berücksichtigt werden?
Auf jeden Fall. Die elektronische Erfassung von Daten und große Schwankungen in ihrer Qualität bilden weiterhin ein großes Hindernis bei der Implementierung. Nicht alle Voruntersuchungen und spezifische Patienteninformationen werden elektronisch festgehalten. Darüber hinaus ist die Beschaffenheit der Daten, von Laborauswertungen zu radiologischen Aufnahmen, sowie die Semantik von Befunden extrem unterschiedlich und dadurch äußerst schwierig, in einem einzigen System abzubilden. Da wartet noch viel Arbeit auf uns.
Abseits der Befundunterstützung forschen Sie auch im Bereich der Risikovorhersagen, welche Rolle spielt hier die medizinische Informatik?
Das IMIBE hat einen großen Schatz an epidemiologischen und genweiten Daten, die wichtige medizinische Erkenntnisse liefern können
Christoph M. Friedrich
Mithilfe von computergestützten Auswertungen können wir gerade im Bereich der genweiten Analyse wichtige Anhaltspunkte für Risikofaktoren und deren Auswirkungen herausfiltern. Eine internationale Studie, bei der ich damals noch am Fraunhofer Institut mitgewirkt habe, hatte beispielsweise das Ziel, Risikogene, die die Entwicklung von intrakraniellen Aneurysmen begünstigen, zu entschlüsseln. Während eine Forschergruppe in Barcelona Bilddaten analysierte und hämodynamische Simulationen erstellte, haben wir in Deutschland via Text Mining nach Risikofaktoren auf genetischer Basis gesucht – mit Erfolg. Wir konnten neben den bekannten assoziierten genetischen Erkrankungen, wie zum Beispiel der polyzystischen Nierenerkrankung (PKD), drei neue Risikofaktoren, sogenannte SNPS (Single Nucleotide Polymorphism) also Genmutationen, identifizieren.
In eine ähnliche Richtung geht auch das neueste gemeinsame Forschungsprojekt des IMIBE und der Fachhochschule. Wir wollen herausfinden, welche Gene oder Genmutationen den Body-Mass-Index eines Patienten in die Höhe treiben. Das IMIBE hat einen großen Schatz an epidemiologischen und genweiten Daten, die wichtige medizinische Erkenntnisse liefern können. Als Informatiker sind wir zudem auch an den größeren Zusammenhängen interessiert. Eine der Thesen, die wir validieren möchten, ist, dass Mutationen in Interaktion treten und gegebenenfalls additiv wirken oder sich gegenseitig aushebeln. In diesem Umfang ist das kein konkretes Projekt, sondern eine bioinformatische Lebensaufgabe.
Vielen Dank für das Gespräch.
Profil:
Prof. Dr.-Ing. Christoph M. Friedrich ist seit 2013 Professor für die Lehrgebiete Informatik und Biomedizinische Informatik an der Fachhochschule Dortmund und seit kurzem auch am Institut für Medizinische Informatik, Biometrie und Epidemiologie (IMIBE) am Universitätsklinikum Essen tätig. Seine Lehr- und Forschungsschwerpunkte liegen in der Medizinstatistik sowie im maschinellen Lernen, aber auch in der Bildverarbeitung und der virtuellen Realität. Temporär betreut er darüber hinaus den Masterstudiengang Medizinische Informatik. Der Diplom-Informatiker hat in Dortmund studiert, bevor er als IT-Manager und Softwareentwickler tätig war. 2005 wurde er Gruppenleiter für Datamining in der Abteilung für Bioinformatik am Fraunhofer Institut für Algorithmen und wissenschaftliches Rechnen (SCAI) in Sankt Augustin bei Bonn. Ein Jahr später erfolgte die Promotion an der Universität Witten/Herdecke. 2010 wurde er Professor für die Lehrgebiete Mathematik für Informatiker, sowie angewandte Informatik an der FH Dortmund, bevor er zur biomedizinischen Informatik wechselte.
Veranstaltungshinweis
Mi, 09.05.18, 13:00-14:30:
Grundlagen von KI-gestützten Diagnosesystemen
C. Friedrich (D-Dortmund)
Wissenschaftliche Sitzung: Forum IT I - Grundlagen und Möglichkeiten KI-gestützter DIagnosesysteme
09.05.2018