Sequenzierungsmethode
Momentaufnahme des Immunsystems
Das Immunsystem jedes einzelnen Menschen kann bis zu mehrere hunderttausend verschiedene T-Zellen umfassen, die sich anhand ihrer Rezeptoren voneinander unterscheiden. Diese enorme Diversität ist die Basis einer schlagkräftigen Immunabwehr. Wissenschaftler aus dem Deutschen Krebsforschungszentrum und dem Nationalen Centrum für Tumorerkrankungen (NCT) Heidelberg entwickelten eine Sequenzierungsmethode, mit der sie das Repertoire an T-Zell-Rezeptoren eines Menschen genau analysieren können. So lässt sich mitverfolgen, wie das Abwehrsystem auf Krankheiten reagiert.
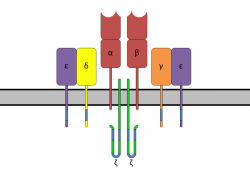
Jeder Mensch ist einzigartig – erst recht auf molekularer Ebene: Jeder 100. bis 300. Baustein im Genom unterscheidet sich von Mensch zu Mensch. Ganz besonders ausgeprägt ist die molekulare Individualität, wenn man die Erbanlagen für die Rezeptoren des Immunsystems betrachtet.
Zu diesen hochvariablen Rezeptoren zählen neben den Antikörpern auch die T-Zell-Rezeptoren. „Diese Diversität der Immun-Rezeptoren ist enorm wichtig, um eine möglichst große Zahl verschiedener körperfremder Substanzen passgenau erkennen zu können“, sagt Manfred Schmidt vom Nationalen Centrum für Tumorerkrankungen (NCT) Heidelberg.
Die extreme Diversität der T-Zellrezeptoren hat eine genetische Grundlage. Jede T-Zelle trägt jedoch ausschließlich Rezeptoren einer einzigen Spezifität. „Die Spezifität und die Häufigkeit der Rezeptor-Gene ergeben zusammengenommen ein genaues Bild des T-Zell-Repertoires des einzelnen Menschen. Aus der Häufigkeit, mit der einzelne T-Zell-Klone vorkommen, können wir Rückschlüsse über Krankheiten und Therapieerfolge ziehen“, so Schmidt.
Die Analyse der hochvariablen Abschnitte der T-Zell-Rezeptor-Gene und die genaue Bestimmung ihrer jeweiligen Häufigkeit stellt eine große Herausforderung dar. Den Wissenschaftlern aus den Arbeitsgruppen von Manfred Schmidt und von Christoph von Kalle, Direktor am NCT, gelang es nun, ein neues Verfahren* zu entwickeln, mit dem präzise Sequenzanalysen des T-Zell -Repertoires gelingen.
Die beiden Erstautoren Eliana Ruggiero und Jan Nicolay erprobten die Methode zunächst an Immunzellen aus dem Blut sechs gesunder Spender. Dort fand sich die erwartete „Polyklonalität“, die Forscher konnten bei jeder Person tausende verschiedener T-Zell-Klone nachweisen.
Wie eine Infektion das T-Zell-Repertoire beeinflusst, ließ sich am Blut von Personen mitverfolgen, die mit dem verbreiteten Cytomegalie-Virus infiziert waren. Diese Blutzellen behandelten die Forscher mit einer Mischung von Peptiden, die verschiedene Antigene des Virus repräsentierten – und simulierten damit den Effekt einer Impfung. Kurz nach Zugabe der Viruspeptide stieg der Anteil derjenigen T-Zellen, die gegen CMV gerichtet waren, und erreichte nach etwa zwei Wochen fast hundert Prozent.
Bei zehn Patienten, die an einem aggressiven T-Zell-Lymphom litten, konnten die Forscher anhand der TCR Untersuchungen im Blut erkennen, wie sehr die Erkrankung das normale T-Zell-Repertoire reduziert.
„Mit dem neuen Vervielfältigungs- und Sequenzierungsverfahren können wir nun beispielsweise bei Kranken mit schweren Infektionen mitverfolgen, wie das Immunsystem mit den Erregern fertig wird“, sagt Christof von Kalle. Auch bei Patienten mit Autoimmunerkrankungen lässt sich so feststellen, welche T-Zell-Klone das körpereigene Gewebe attackieren, bei Lymphompatienten können Ärzte den Stand der bösartigen Erkrankung mitverfolgen.“
Besonders vielversprechend für Krebsmediziner wie von Kalle ist der Einsatz des Verfahrens für die Tumor-Immuntherapie: Sie können nun die DNA-Sequenz der wirksamsten gegen den Tumor gerichteten T-Zell-Rezeptoren ermitteln und möglicherweise künstlich nachbauen. Der immense Fortschritt bei der Sequenzierung von DNA und RNA – da sind sich die Autoren einig – wird zukünftig noch effizientere Einblicke in das menschliche Immunsystem erlauben.
* TCR ligation anchored-magnetically captured PCR (TCR-LA-MC PCR)
Quelle: Deutsches Krebsforschungsinstitut
02.09.2015