Quelle: HZI / Manfred Rohde
News • Infektionsforschung
Ein Lichtblick für die mikrobiologische Diagnostik
Der diagnostische Nachweis von bakteriellen Krankheitserregern in klinischen Proben ist eine wichtige Voraussetzung für die erfolgreiche antimikrobielle Behandlung. Ein Team des Helmholtz-Zentrums für Infektionsforschung (HZI), des Deutschen Zentrums für Infektionsforschung (DZIF) und der Tel Aviv University, Israel, hat nun molekulare Sonden entwickelt, die verschiedene Bakterienspezies hochsensitiv nachweisen können. Bakterielle Zellen aktivieren die Sonden und bringen sie zum Leuchten. Dieses Signal ermöglicht den Erregernachweis direkt in klinischen Proben.
In Anbetracht der steigenden Gefahr durch Antibiotikaresistenzen sollten Erkrankungen nicht auf Verdacht mit Antibiotika behandelt werden. Zum verantwortungsvollen Einsatz von Antibiotika gehört daher der vorhergehende Nachweis, dass tatsächlich eine bakterielle Infektion vorliegt. Mikrobiologische Methoden, bei denen die Erreger auf Nährmedien angezüchtet werden, nehmen jedoch viel Zeit in Anspruch und gelingen oft nicht, wenn Patient:innen beispielsweise bereits mit Antibiotika behandelt wurden. Daher ist eine verbesserte Diagnostik ein wichtiges Element im Kampf gegen Antibiotikaresistenzen.
Dazu will ein Forschungsteam unter der Leitung von Prof. Mark Brönstrup vom HZ) nun mit neuartigen Sonden beitragen. Die Forscher:innen machen sich eine Molekülklasse zu eigen, mit der Bakterien eisenhaltige Verbindungen aufnehmen – die Siderophore. „Eisen ist im Körper ein knappes Gut. Daher nehmen Bakterien auch Siderophore auf, die chemisch nicht genau denen entsprechen, die sie selber herstellen“, sagt Carsten Peukert, Doktorand in der Abteilung „Chemische Biologie“ am HZI und Erstautor der Studie. „Das prädestiniert Siderophore für den Einsatz als molekulare trojanische Pferde.“
An die Siderophore koppelten die Forscher:innen ein Molekül der Gruppe Dioxetane. Mit Dioxetanen verwandte Moleküle sind auch in Glühwürmchen für das charakteristische Leuchten verantwortlich. Eine externe Lichtquelle ist dafür nicht notwendig. Damit die Sonden jedoch nur spezifisch in Bakterien leuchten, integrierten die Forscher:innen einen molekularen Lichtschalter. Dieser wird betätigt, wenn Bakterien die Sonden in ihrem Zellinneren enzymatisch spalten.
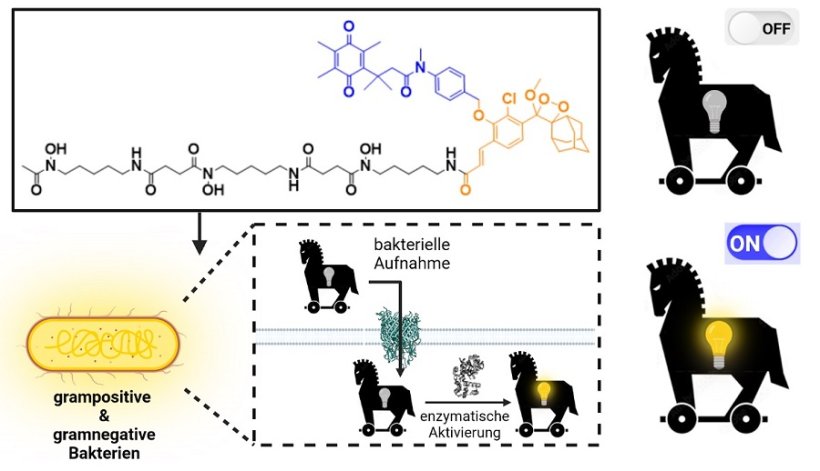
Quelle: HZI/Carsten Peukert
Das Forschungsteam kann mit seiner Sonde alle ESKAPE-Pathogene nachweisen. Der Begriff fasst die Erreger Enterococcus faecium, Staphylococcus aureus, Klebsiella pneumoniae, Acinetobacter baumannii, Pseudomonas aeruginosa und Enterobacter-Spezies zusammen. Diese Erreger gelten als typische Vertreter der Krankenhauskeime und sind besonders häufig resistent gegen Antibiotika. „Besonders gramnegative Bakterien wie Pseudomonaden oder Klebsiellen, die über zwei Zellmembranen verfügen, konnten bisher mit den Vorläufern unserer Sonden nicht zuverlässig markiert werden. Wir haben das System nun so weiterentwickelt, dass auch diese Gruppe nachweisbar ist“, sagt Studienleiter Mark Brönstrup, Leiter der Abteilung „Chemische Biologie“ am HZI und Wissenschaftler am DZIF im Forschungsbereich „Neue Antibiotika“. Die Sonden können auch Bakterien nachweisen, die sich innerhalb von Wirtszellen vermehren. Die technischen Anforderungen zum Nachweis der Sonden in klinischen Proben wie Blutplasma sind vergleichsweise niedrig. Daher könnte das System zukünftig auch abseits spezialisierter Labore zum Einsatz kommen.
Andere molekulare Diagnostikmethoden wie die Massenspektrometrie oder die Polymerasekettenreaktion unterscheiden nicht zwischen toten und lebenden bakteriellen Zellen. „Unsere Methode bietet den Vorteil, dass nur lebende Bakterien nachgewiesen werden. Wenn die Sonden aufleuchten, liegt also eine aktive Infektion vor“, sagt Brönstrup. Neben dem Einsatz in klinischen Proben sei auch der Einsatz in der Lebensmittelhygiene denkbar. Zudem könne das Siderophor-basierte System auch angepasst werden und anstatt der leuchtenden Sonden etwa antimikrobielle Wirkstoffe einschleusen. Der Lichtblick in der Diagnostik soll also nur der Anfang der Anwendungen für die molekularen trojanischen Pferde der HZI-Forscher:innen sein.
Die Forschungsergebnisse wurden in der Fachzeitschrift Angewandte Chemie veröffentlicht.
Quelle: Helmholtz-Zentrum für Infektionsforschung
13.05.2022