© Rajewsky Lab, Max Delbrück Center
News • Räumliche Transkriptomik und ECM-Bildgebung
3D-Einblicke in die Tumor-Umgebung
Eine Kombination hochauflösender räumlicher Einzelzelltechnologien kann die zelluläre Nachbarschaft eines Tumors in 3D kartieren und so Angriffspunkte für eine personalisierte Krebstherapie finden.
Das schreibt ein Team um Nikolaus Rajewsky vom Max Delbrück Center in zwei „Cell Systems“-Studien.1,2
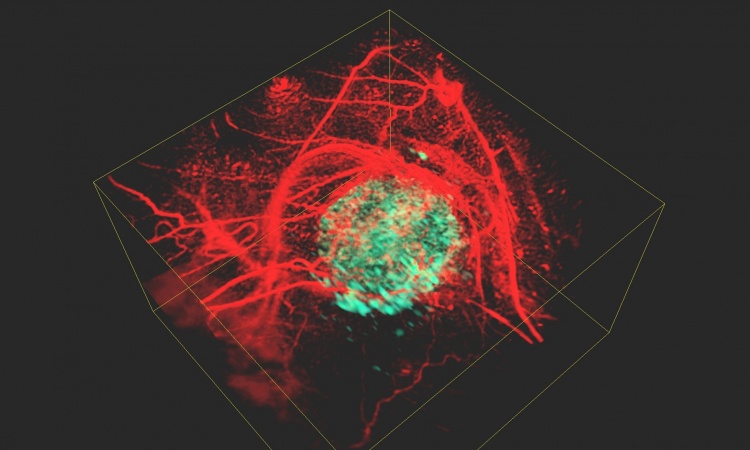
Es geht nicht nur darum, welche Zellen in einem Tumor vorkommen. Wirklich detailliert wird das Bild erst dann, wenn man versteht, wo sie sich genau befinden und wie sie mit anderen Zellen in ihrer Umgebung interagieren. Solche Einblicke in das Zusammenspiel innerhalb der Zellnachbarschaften könnten Ärzten in Zukunft helfen, eine für den jeweiligen Patienten besonders wirksame Therapie auszuwählen. Ein Forschungsteam vom Berliner Institut für Medizinische Systembiologie des Max Delbrück Center (MDC-BIMSB) hat jetzt gemeinsam mit internationalen Kollegen Technologien der räumlichen Transkriptomik mit Bildern der extrazellulären Matrix kombiniert. So konnten sie das Innenleben eines Lungentumors im Frühstadium in beispielloser Präzision beschreiben. Die „Proof-of-Concept“-Studien sind in „Cell Systems“ publiziert.
Wir wissen also nicht nur, welche Zelltypen vorhanden sind. Wir sehen auch, wie sie sich mit ihren Nachbarn gruppieren, und beginnen zu verstehen, wie Krebszellen das gesunde Gewebe um sie herum dazu bringen, das Tumorwachstum zu unterstützen
Tancredi Massimo Pentimalli
„Tumore sind komplexe Ökosysteme. Dort leben Tumorzellen in enger Nachbarschaft mit der sie umgebenden extrazellulären Matrix“, sagt Professor Nikolaus Rajewsky, der Direktor des MDC-BIMSB und Letztautor beider Studien. „Die Daten, die wir inzwischen aus dem Tumorgewebe eines Patienten gewinnen können, sind so präzise und umfassend, dass wir auf dieser Grundlage die molekularen Mechanismen des Phänotyps rechnerisch vorhersagen können. Das ist wirklich neu – und von fundamentaler Bedeutung, wenn die personalisierte Medizin Wirklichkeit werden soll.“
Die Transkriptomik dokumentiert, welche RNA die Zellen zu einem bestimmten Zeitpunkt aktiv nutzen. Das gibt zum einen Hinweise darauf, was diese Zellen gerade machen. Zum anderen zeigt es, welche Zelltypen in einer Probe vorhanden sind. In der räumlichen Transkriptomik wird zunächst für die einzelnen Zellen eine 2D-Karte erstellt. Rajewsky und seine Kollegen konnten dafür ein neues Instrument der Firma NanoString namens CosMx nutzen. Es arbeitet mit einer besonders hohen Auflösung und kann tausend verschiedene RNA-Moleküle gleichzeitig detektieren – traditionelle Methoden spüren eine Handvoll Moleküle gleichzeitig auf. Das Team analysierte so 340.000 einzelne Zellen des Lungentumors und fand dabei 18 Zelltypen.
Ein neuer Algorithmus, STIM genannt, ermöglichte anschließend die 3D-Kartierung. Er rekonstruiert aus den Daten der 2D-Analyse virtuelle 3D-Gewebeblöcke. „Wir hatten die Idee, die Datensätze der räumlichen Transkriptomik als Bilder zu modellieren“, sagt Dr. Nikos Karaiskos, Postdoktorand in der AG von Nikolaus Rajewsky und Co-korrespondierender Autor des zweiten „Cell Systems“-Papers, das STIM im Detail beschreibt. Mithilfe bildgebender Technologien gelingt es STIM, maschinelles Sehen und räumliche Transkriptomik zu verbinden. Das Team arbeitete dabei eng mit Dr. Stephan Preibisch zusammen, der Gruppenleiter am MDC-BIMSB war, inzwischen aber zum Howard Hughes Medical Institute’s Janelia Research Campus in den USA gewechselt ist.
Um mit einem weiteren Bildgebungsverfahren namens Second Harmonic Generation Imaging Elastin und Kollagen, die Hauptbestandteile der extrazellulären Matrix in der Lunge, in den zellulären Nachbarschaften zu kartieren, kooperierten die Forschenden um Rajewsky mit der Technologieplattform „Systems Biology Imaging“ am MDC-BIMSB. Wie das Team herausfand, waren Bereiche mit mehr Elastin gesünder. Tumorzellen hingegen waren von mehr Kollagen umgeben. Dies deutet den Wissenschaftlern zufolge darauf hin, dass sich das Gewebe dort auf schädliche Art und Weise umbaut.
„Wir wissen also nicht nur, welche Zelltypen vorhanden sind. Wir sehen auch, wie sie sich mit ihren Nachbarn gruppieren, und beginnen zu verstehen, wie Krebszellen das gesunde Gewebe um sie herum dazu bringen, das Tumorwachstum zu unterstützen“, sagt Tancredi Massimo Pentimalli, Arzt und Doktorand in der AG Rajewsky sowie an der Berlin School of Integrative Oncology der Charité – Universitätsmedizin Berlin. Er ist Erstautor des ersten Papers.
Das ist Pathologie 2.0 – wir schauen uns die Zellen nicht einfach nur unter dem Mikroskop an, um eine Diagnose zu stellen, sondern bringen umfassende molekulare Einblicke in die Klinik
Tancredi Massimo Pentimalli
Die Analyse ging noch weiter. Unter anderem konnten die Forschenden prüfen, ob Fibroblasten, die Bindegewebe bilden, im Tumor aktiv waren und das Gewebe umbauten. Darüber hinaus belauschten sie die Kommunikation der Zellen untereinander und erkannten dabei, wie die Krebszellen verhinderten, dass Immunzellen in den Tumor eindringen konnten. „Dieser Mechanismus der Immunsuppression ist bekannt und legt nahe, dass eine Immuntherapie hier erfolgversprechend gewesen wäre“, sagt Pentimalli: „Checkpoint-Inhibitoren können die Bremse lösen und dann kann eine ganze Armada von Immunzellen eingreifen. Es war spannend zu sehen, dass unser Ansatz diese Dynamik aufdecken und einen personalisierten Ansatz einer Immuntherapie ermöglichen kann.“ Diese entscheidenden Einblicke waren nur in 3D möglich. Mit 2D-Daten konnten die Forschenden die Krebszellen nicht von den in die Tumoroberfläche eingebetteten Immunzellen unterscheiden.
Der Clou an dem High-Tech-Ansatz: Ausgangspunkt ist eine einfache Gewebeprobe, wie sie in jedem Pathologie-Labor zu finden ist. Für die vorliegenden Studien nutzten die Forschenden eine Probe aus einem Lungentumor, der seit mehreren Jahren in Formalin konserviert und in Paraffin-Wachs eingebettet war. Mit dieser Standardmethode archivieren Pathologen Gewebe. „Wir konnten diese Fülle molekularer Informationen gewinnen, obwohl wir mit einem sehr dünnen Gewebeschnitt gearbeitet haben und die Probe seit Jahren bei Raumtemperatur gelagert worden war“, sagt Pentimalli. „Das ist Pathologie 2.0 – wir schauen uns die Zellen nicht einfach nur unter dem Mikroskop an, um eine Diagnose zu stellen, sondern bringen umfassende molekulare Einblicke in die Klinik.“
Das Team will den Ansatz nun auf größere Datensätze anwenden. Derzeit arbeiten die Forschenden an 700 Tumorproben von 200 Patienten und wollen gemeinsam mit Dr. Fabian Coscia, der die AG „Räumliche Proteomik“ am Max Delbrück Center leitet, auch die Ebene der Proteine in ihre Analyse integrieren.
Quelle: Max Delbrück Center; Text: Laura Petersen
03.05.2025