DNA
Zwischen den Genen lesen
Unsere Gene entscheiden über vieles in unserem Leben – wie wir aussehen, welche Begabungen wir haben oder welche Krankheiten wir entwickeln. Lange Zeit als „Müll“ abgetan, weiß man inzwischen, dass auch DNA-Bereiche zwischen den Genen wichtige Funktionen haben. Sie sind Sitz eines komplexen Steuerungswerks mit Tausenden von molekularen Schaltern, die die Aktivität unserer Gene regulieren. Doch die regulatorischen Bereiche des Erbguts lassen sich schwer finden. Forscher um Patrick Cramer vom MPI für biophysikalische Chemie und Julien Gagneur von der TU München haben jetzt eine Methode entwickelt, mit der sich regulatorische DNA-Bereiche aufspüren lassen, die aktiv sind und Gene steuern.
Die Gene in unserer DNA enthalten die Baupläne für Proteine, die als „Arbeiter“ praktisch alle Prozesse in unseren Zellen ausführen und steuern. Doch damit jedes Protein zur rechten Zeit am rechten Ort in unserem Körper seine Aufgaben erfüllen kann, muss die Aktivität des dazugehörigen Gens genau kontrolliert werden. Diese Funktion übernehmen regulatorische DNA-Bereiche zwischen den Genen, die als hochkomplexes Steuerungswerk fungieren. „Regulatorische DNA-Bereiche sind unter anderem lebenswichtig für die Entwicklung des Menschen, den Erhalt von Geweben und die Immunantwort“, erklärt Patrick Cramer, Leiter der Abteilung Molekularbiologie am Max-Planck-Institut für biophysikalische Chemie. „Darüber hinaus spielen sie bei vielen Krankheiten eine wichtige Rolle. Krebs- und Herzkreislaufpatienten bespielsweise haben genau in diesen DNA-Abschnitten viele Veränderungen“, so der Biochemiker.
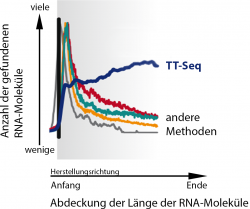
Wenn regulatorische DNA-Bereiche aktiv sind, werden von ihnen zunächst RNA-Kopien erstellt. „Die daraus resultierenden RNA-Moleküle haben für uns Forscher allerdings einen großen Nachteil: Sie werden von der Zelle rasch wieder abgebaut und lassen sich daher bislang nur schwer aufspüren“, berichtet Julien Gagneur, der vor Kurzem mit seiner Gruppe vom Genzentrum der Ludwig-Maximilians-Universität München an die Technische Universität München gewechselt ist. „Aber gerade die sehr kurzlebigen RNA-Moleküle wirken oft als lebenswichtige molekulare Schalter, die Gene gezielt aktivieren, wenn sie an einem bestimmten Ort des Körpers benötigt werden. Ohne diese Schalter würden unsere Gene nicht funktionieren.“
Anker für kurzlebige molekulare Schalter
Björn Schwalb und Margaux Michel, Mitarbeiter in Cramers Team, ist es gemeinsam mit Benedikt Zacher Gagneurs Gruppe nun gelungen, eine hochempfindliche Methode zu entwickeln, mit der sich auch sehr kurzlebige RNA-Moleküle einfangen und identifizieren lassen – das sogenannte TT-Seq (für englisch: transient transcriptome sequencing). Um die RNA-Moleküle einzufangen, verwendeten die drei Nachwuchsforscher einen Trick: Sie verabreichten den Zellen für einige Minuten ein Molekül, das als eine Art Anker wirkt. Die Zellen bauten daraufhin diesen Anker in jede RNA ein, die sie in der Versuchszeit herstellten. Mithilfe des Ankers konnten die Wissenschaftler schließlich auch die kurzlebigen RNA-Moleküle aus der Zelle herausfischen und untersuchen.
„Die so gefundenen RNA-Moleküle stellen eine Momentaufnahme aller DNA-Bereiche dar, die zu einem bestimmten Zeitpunkt in der Zelle aktiv waren – der Gene ebenso wie der bislang schwer auffindbaren regulatorischen Bereiche zwischen den Genen“, erläutert Cramer. „Mit der TT-Seq-Methode haben wir jetzt das geeignete Werkzeug an der Hand, um etwas darüber zu lernen, wie Gene in verschiedenen Zelltypen gesteuert werden und wie genregulatorische Programme arbeiten,“ ergänzt Gagneur.
In vielen Fällen haben Forscher schon eine recht genaue Vorstellung davon, welche Gene bei einer bestimmten Krankheit eine Rolle spielen, kennen aber die daran beteiligten molekularen Schalter nicht. Die Wissenschaftler um Cramer und Gagneur hoffen, mithilfe der neuen Methode dazu beitragen zu können, wichtige Schlüsselmechanismen aufzudecken, die bei der Entstehung und dem Verlauf von Krankheiten eine Rolle spielen. In einem nächsten Schritt möchten sie ihre Methode unter anderem auf Blutzellen anwenden, um den Verlauf einer HIV-Infektion bei AIDS-Patienten besser zu verstehen.
Original-Publikation
Björn Schwalb, Margaux Michel, Benedikt Zacher, Katja Frühauf, Carina Demel, Achim Tresch, Julien Gagneur, Patrick Cramer: TT-Seq maps the human transient transcriptome. Science 352,1225-1228 (2016), doi: 10.1126/science.aad9841.
Quelle: Max-Planck-Institut für biophysikalische Chemie
27.06.2016