News • Translation und Transkription
Proteinbiosynthese: Neue Perspektiven für die Antibiotika-Forschung
Forscher der Universität Bayreuth und der Columbia University in New York berichten über wegweisende Erkenntnisse zur Proteinbiosynthese in Bakterien.
Das kleine Protein NusG verknüpft zwei große molekulare Maschinen, die bei der Genexpression, der Herstellung bakterieller Proteine auf der Basis von Erbinformationen, zusammenwirken: die RNA-Polymerase und das Ribosom. Die molekulare Brücke versetzt die Bakterienzelle in die Lage, die zeitlich aufeinander folgenden Abschnitte der Genexpression, die Transkription und die Translation, optimal aufeinander abzustimmen. Sie könnte deshalb ein hervorragender Angriffspunkt für künftige antibiotische Wirkstoffe sein.
Die Wissenschaftler veröffentlichten ihre Erkenntnisse jetzt in der Zeitschrift „iScience“.
Grafik: Philipp Zuber
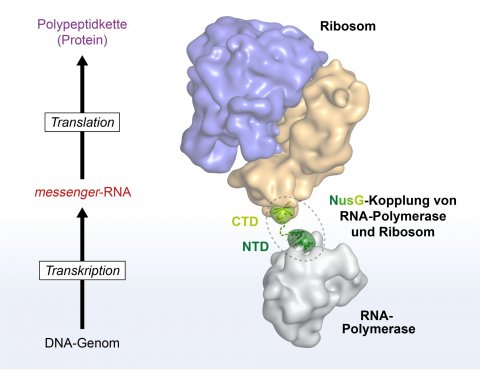
In allen Lebewesen ist die Genexpression ein zweistufiger Prozess: Zunächst werden die in der DNA gespeicherten Erbinformationen als Vorlage genutzt, um auf dieser Basis Ribonukleinsäuren, sogenannte messenger-RNAs (mRNAs), zu synthetisieren. Dadurch werden die Erbinformationen in eine für die Zelle unmittelbar verwertbare Form gebracht. Für diesen Vorgang, die Transkription, ist die RNA-Polymerase zuständig. Die messenger-RNAs wiederum enthalten die molekularen Baupläne, die vom Ribosom erkannt und für die Herstellung entsprechender Proteine, die Translation, genutzt werden. Bei Menschen und Tieren sind diese beiden Abschnitte der Genexpression räumlich und biochemisch klar getrennt. In den Zellen von Bakterien sind sie hingegen, wie man schon seit mehr als 50 Jahren weiß, aneinander gekoppelt.
Bereits vor zehn Jahren publizierte eine Bayreuther Forschungsgruppe unter der Leitung von Prof. Dr. Paul Rösch in „Science“ erste Indizien dafür, dass die Kopplung durch das Protein NusG verursacht sein könnte. Doch erst jetzt gelang der Forschungsgruppe von Dr. Stefan H. Knauer in Zusammenarbeit mit Partnern an der Columbia University, New York, der erste, direkte strukturelle Nachweis. NusG besteht aus zwei flexibel verbundenen Bereichen: einer aminoterminalen Domäne (NTD) und einer carboxyterminalen Domäne (CTD). Die CTD bindet an das Ribosom, die NTD an die RNA-Polymerase. Auf diese Weise bildet NusG eine flexible Brücke zwischen den zentralen Maschinen der Genexpression, ähnlich einer beweglichen Kupplung zwischen Eisenbahnwaggons. Diese Verbindung bewirkt, dass Transkription und Translation zeitlich aufeinander abgestimmt sind. Experimente mit hochauflösender magnetischer Kernresonanz-Spektroskopie (NMR), die am Nordbayerischen NMR-Zentrum der Universität Bayreuth durchgeführt wurden, haben diese Zusammenhänge eindeutig sichtbar gemacht.

Foto: Rainer Hofmann
„Damit eröffnen sich hochinteressante Perspektiven für die Entwicklung antibiotischer Wirkstoffe. Wenn es gelingt, diesen molekularen Brückenbau zu verhindern, könnte die bakterielle Proteinsynthese und damit auch die Vermehrung von Bakterien empfindlich gestört werden – und zwar ohne dass der menschliche Organismus dadurch beeinträchtigt wird. Wir konnten in dieser Hinsicht schon erste vielversprechende Forschungsergebnisse erzielen“, sagt Dr. Stefan Knauer. „Der Nachweis für die zentrale Rolle von NusG bei der bakteriellen Proteinbiosynthese ist uns vor allem dadurch gelungen, dass wir strukturbiologische, biochemische und molekularbiologische Verfahren miteinander kombiniert haben. Diese interdisziplinäre Herangehensweise wollen wir auch bei der Suche nach effizienten Wirkstoffen weiter verfolgen“, ergänzt Mitautor Philipp Zuber M.Sc., der an der Universität Bayreuth promoviert und hier das Elitestudienprogramm "Macromolecular Science" im Rahmen des Elitenetzwerks Bayern absolviert hat.
Die Studie ist hervorgegangen aus einer engen Zusammenarbeit der Bayreuther Forscher mit den Arbeitsgruppen von Prof. Dr. Max Gottesman und Prof. Dr. Joachim Frank an der Columbia University in New York. Frank erhielt 2017 den Chemie-Nobelpreis für die Weiterentwicklung der Kryoelektronenmikroskopie, einer Forschungstechnologie, die auch bei der neuen Studie zum Einsatz kam.
Quelle: Universität Bayreuth
21.07.2020