Bildquelle: DPZ/Markus Hoffmann
News • Mechanismus entschlüsselt
Forscher klären Aktivierung von SARS-CoV-2
Das SARS-Coronavirus 2 (SARS-CoV-2) infiziert Lungenzellen und ist für die Covid-19-Pandemie verantwortlich. Das sogenannte Spike-Protein dient dem Virus als Schlüssel zur Wirtszelle und trägt eine ungewöhnliche Aktivierungssequenz.
Die Abteilung Infektionsbiologie des Deutschen Primatenzentrums (DPZ) – Leibniz-Institut für Primatenforschung hat jetzt gezeigt, dass diese Sequenz durch das zelluläre Enzym Furin gespalten wird und dass die Spaltung für die Infektion von Lungenzellen wichtig ist. Diese Ergebnisse definieren neue Ansatzpunkte für Therapie und Impfstoffforschung. Außerdem geben sie Hinweise darauf, wie sich Coronaviren von Tieren verändern müssen, um sich in der menschlichen Bevölkerung ausbreiten zu können.
Die Wissenschaftler veröffentlichten ihre Erkenntnisse jetzt im Fachjournal Molecular Cell.
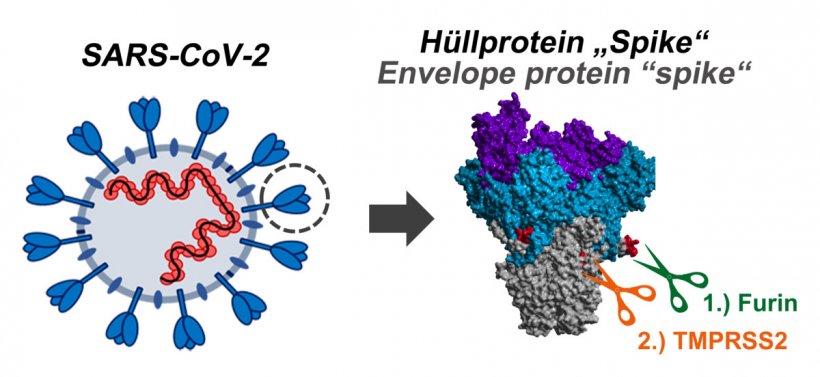
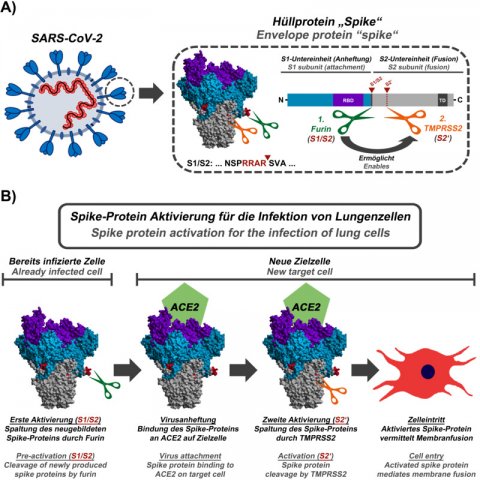
Das neue Coronavirus SARS-CoV-2 wurde von Tieren auf den Menschen übertragen und breitet sich weltweit aus. Es verursacht die neue Lungenkrankheit Covid-19, an der schon über 200.000 Menschen gestorben sind. Das Spike-Protein auf der Virusoberfläche dient dem Virus als Schlüssel zur Wirtszelle. Es erlaubt dem Virus, sich an Zellen anzuheften und anschließend mit den Zellen zu verschmelzen, um so seine Erbinformation für die virale Vermehrung in die Zellen einzuschleusen. Dazu müssen Aktivierungssequenzen des Spike-Proteins durch Enzyme der Zellen, sogenannte Proteasen, gespalten werden. Das Spike-Protein von SARS-CoV-2 trägt eine Aktivierungssequenz an der sogenannten S1/S2-Spaltstelle, die man von hochpathogenen Vogelgrippe-Viren kennt, aber in SARS-CoV-2 eng verwandten Viren bislang nicht finden konnte. Die Bedeutung dieser Sequenz für das Virus war bisher unbekannt.
Bildquelle: DPZ/Markus Hoffmann
Die Infektionsbiologen des Deutschen Primatenzentrums um Markus Hoffmann und Stefan Pöhlmann konnten in ihrer aktuellen Studie zeigen, dass die S1/S2-Aktivierungssequenz des Spike-Proteins von SARS-CoV-2 durch die zelluläre Protease Furin gespalten wird. Diese Spaltung ist für die Infektion von Lungenzellen unverzichtbar. Außerdem ist sie für die Verschmelzung von infizierten Zellen mit nicht-infizierten Zellen wichtig, ein weiterer Weg, wie sich das Virus im Körper ausbreiten könnte, ohne die Wirtszelle zu verlassen. „Unsere Ergebnisse deuten darauf hin, dass die Hemmung von Furin die SARS-CoV-2-Ausbreitung in der Lunge blockieren sollte“, sagt Stefan Pöhlmann, Leiter der Abteilung Infektionsbiologie am DPZ. „Außerdem demonstrieren sie, zusammen mit unseren vorangegangenen Arbeiten, dass das Virus einen zweistufigen Aktivierungsmechanismus nutzt: In infizierten Zellen muss das Spike-Protein durch die Protease Furin gespalten werden, damit neu gebildete Viren anschließend die Protease TMPRSS2 für eine weitere Spaltung des Spike-Proteins nutzen können, die für das Eindringen in Lungenzellen wichtig ist.“

Bildquelle: DPZ/Karin Tilch
Damit ein Lebendimpfstoff eine starke Immunantwort auslöst, muss er sich in begrenztem Maße im Körper vermehren können, beispielsweise lokal am Ort der Injektion. „SARS-CoV-2-Varianten, bei denen die Aktivierungssequenz für Furin entfernt wurde, könnten als Basis für die Entwicklung von solchen Lebendimpfstoffen genutzt werden, da die fehlende Spaltung des Spike-Proteins die Virus-Ausbreitung im Körper stark einschränken sollte. Ein hinreichend abgeschwächtes Virus würde nicht mehr in der Lage sein, Krankheitssymptome auszulösen, dem Immunsystem aber dennoch ermöglichen, auf den Erreger zu reagieren und beispielsweise neutralisierende Antikörper zu bilden“, sagt Markus Hoffmann, Erstautor der Studie.
In Wildtieren, insbesondere Fledermäusen, wurde in den vergangenen 20 Jahren eine Vielzahl von Coronaviren entdeckt, die eine enge Verwandtschaft zum SARS-CoV und SARS-CoV-2 besitzen. Allerdings wurde bislang nur bei SARS-CoV-2 eine S1/S2-Aktivierungssequenz beobachtet, die von Furin gespalten werden kann. „Die Beprobung von Wildtieren und die gezielte Suche nach Coronaviren mit einem Fokus auf die S1/S2-Aktivierungssequenz ist notwendig, um solche Viren zu identifizieren, für die das Risiko besteht, dass sie den Menschen infizieren und effizient verbreitet werden können. Außerdem sollte man bei eventuellen zukünftigen Coronavirus-Ausbrüchen spezifisch die Sequenz der S1/S2-Spaltstelle analysieren, da sie als Marker für Übertragbarkeit dienen könnte“, sagt Markus Hoffmann.
Quelle: Deutsches Primatenzentrum (DPZ) – Leibniz-Institut für Primatenforschung
05.05.2020