Warum Nervenzellen sterben
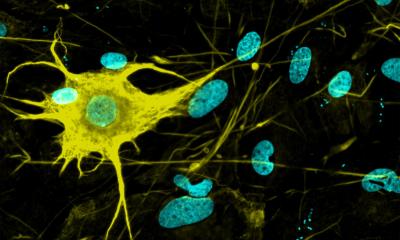
In Hirnzellen von Patienten mit der Alzheimer- oder Huntington-Krankheit können Mediziner und Forscher unter dem Mikroskop Proteinverklumpungen, auch Aggregate genannt, sehen. Dass diese Aggregate zum Tod der Nervenzellen beitragen, wird schon lange vermutet. Wie Science jetzt berichtet, haben Forscher um Mark Hipp und Ulrich Hartl vom Max-Planck-Institut für Biochemie in Martinsried gezeigt, dass der Ort der Aggregate innerhalb der Zelle ihr Überleben beeinflusst. Während Proteinklumpen im Zellkern die Zellfunktion kaum beeinflussen, stören sie im Zellplasma wichtige Transportwege zwischen Zellplasma und Zellkern. Dies führt langfristig zum Tod der Zellen und Voranschreiten der Krankheit.
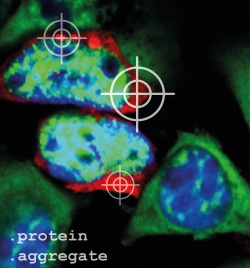
roteine bestehen aus langen Aminosäureketten und funktionieren in Zellen wie kleine Maschinen. Um ihre Arbeit aufnehmen zu können, müssen die Proteine eine vorgegebene dreidimensionale Struktur annehmen. In gesunden Zellen gibt es eine Vielzahl von Faltungshelfern und eine umfangreiche Qualitätskontrolle. Falsch gefaltete Proteine werden entweder repariert oder schnell abgebaut. Geschieht dies nicht oder nicht ausreichend, können Proteine mit sich selbst oder anderen Proteinen zu Aggregaten verklumpen und die Zellen schädigen.
Bei neurodegenerativen Krankheiten wie Alzheimer, Parkinson, ALS und Huntington scheinen solche Proteinaggregate für das Absterben von Nervenzellen mit verantwortlich zu sein. Wie diese Verklumpungen die Zellen schädigen ist bis heute nicht geklärt. Deshalb wurde 2013 das ToPAG-Konsortium (www.topag.mpg.de) ins Leben gerufen, ein Zusammenschluss verschiedener Expertengruppen, die diesem Rätsel auf der Spur sind. Erste Erfolge können jetzt vermeldet werden. So zeigen Wissenschaftler im Labor von Ulrich Hartl, ein weltweit bekannter Experte für Proteinfaltung, dass es für das Überleben der Zelle entscheidend ist, wo sich die Aggregate innerhalb der Zelle befinden.
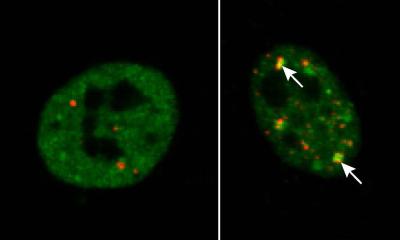
Um dies herauszufinden haben die Forscher, zusammen mit den Forschungsgruppen von Konstanze Winklhofer und Jörg Tatzelt an der Ruhr-Universität Bochum, ein künstlich hergestelltes Protein und das für die Huntington Krankheit verantwortliche Protein Huntingtin in Zellkulturen getestet. Beide Proteine lagern sich von allein zu großen Proteinklumpen zusammen. „Interessanterweise bildet dasselbe Protein im Zellplasma besser lösliche, aber für die Zelle giftigere Aggregate als im Zellkern“, erklärt Mark Hipp, Forschungsgruppenleiter in der Abteilung von Ulrich Hartl und Leiter der Studie. Proteinverklumpungen im Zellplasma verhinderten den Transport von RNA und richtig gefalteten Proteinen zwischen Zellkern und Zellplasma. Weil die Aggregate klebrige Eigenschaften haben, werden aus der Zelle lebensnotwendige Proteine weggefangen. „Wir haben in den Aggregaten im Zellplasma wichtige Bestandteile der zellulären Transportmaschinerie gefunden. Das hat zu Folge, dass die Bestandteile für einen funktionierenden Kerntransport dann fehlen, ungefähr so, als wenn Teile einer Maschine fehlen. Dann kann diese auch nicht im Ganzen funktionieren. Vermutlich ist das die Ursache für den geschädigten Transportweg“, erklärt Andreas Wörner, Erstautor der Studie. Wenn die Bauanleitung der Proteine, die RNA, aus dem Zellkern nicht in das Zellplasma gelangen kann, können dort auch keine Proteine mehr hergestellt werden und die Zelle geht zugrunde. Warum die Aggregate, die sich direkt im Zellkern befinden die Nervenzellen weniger schädigen kann nur vermutet werden. Laut Studie scheint das Kernprotein NPM1 dabei eine zentrale Schutzfunktion auszuüben.
„Die Ergebnisse der Studie bringen uns Forscher und Mediziner ein großes Stück weiter“, fasst Mark Hipp zusammen. „Denn wenn wir wissen, welche Schäden die Aggregate verursachen, können wir in Zukunft passendere Gegenmaßnahmen entwickeln.“
Originalpublikation:
A. C. Woerner, F. Frottin, D. Hornburg, L. R. Feng, F. Meissner, M. Patra, J. Tatzelt, M. Mann, K. F. Winklhofer, U. Hartl, M. S. Hipp: Cytoplasmic protein aggregates interfere with nucleo-cytoplasmic transport of protein and RNA. Science, 8. Januar 2016
DOI: 10.1126/science.aad2033
Quelle: Max-Planck-Institut für Biochemie
19.01.2016