Leuchtende Marker für die Nanoskopie
Wissenschaftler des Göttinger Exzellenzclusters CNMPB und des EMBL in Heidelberg identifizieren effizienten Fluoreszenzmarker für super-auflösende Mikroskopie-Verfahren.
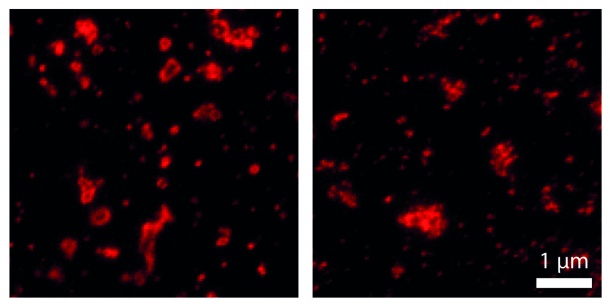
Modernste optische Technologien, wie die super-auflösende Nanoskopie, liefern faszinierende Einblicke in das Innere lebender Zellen. Kleinste Strukturen und molekulare Abläufe lassen sich exakt darstellen. Um diese Vorgänge sichtbar zu machen, müssen die zu untersuchenden Objekte genau markiert werden. Bisher war fraglich, ob die in der konventionellen Bildgebung zur Markierung und Visualisierung eingesetzten fluoreszierenden Proteine auch für den Einsatz in der super-auflösenden Mikroskopie geeignet sind.
Wissenschaftler des Göttinger Exzellenzclusters und DFG-Forschungszentrums für Mikroskopie im Nanometerbereich und Molekularphysiologie des Gehirns (CNMPB) und des European Molecular Biology Laboratory (EMBL) in Heidelberg haben bekannte fluoreszierende Proteine untersucht, ob sie für die Nanoskopie anwendbar sind. Bekannt ist, dass viele Markerproteine aufgrund ihrer relativ großen Größe dazu neigen, Aggregate zu bilden, die Artefakte in nanoskopischen Messungen hinterlassen können. Deshalb interessierten sich die Forscher besonders für die Frage, ob sich die Markierung auf die strukturelle Organisation der Proteine auswirkt, die mit Hilfe der Nanoskopie dargestellt werden sollen.
Die Erkenntnisse des Forscherteams unter der Leitung von Prof. Dr. Silvio O. Rizzoli, Direktor des Instituts für Neuro- und Sinnesphysiologie, Universitätsmedizin Göttingen, zeigen: Fluoreszierende Proteine sind als Mittel zur Markierung in der Nanoskopie und für die biomedizinische Forschung gut geeignet und generell anwendbar. Insbesondere die auf dem Einsatz unnatürlicher Aminosäuren zugrunde liegende Fluoreszenz-Markierung gilt als effiziente Methode. Die Forschungsergebnisse wurden in der Fachzeitschrift ACS Nano veröffentlicht.
„Genetisch kodierte, also durch Genmanipulation in lebende Zellen eingeschleuste Fluoreszenzmarker sind für die Untersuchung molekularer Strukturen und Prozesse besonders interessant, weil sie einfach zu handhaben und für die Zellen gut verträglich sind“, sagt Prof. Rizzoli.
Die Wissenschaftler konzentrierten sich auf insgesamt 26 Proteine, von denen bekannt ist, dass sie zur Bildung multimolekularer Aggregate neigen. Die Forscher verglichen die strukturelle Organisation der Proteine mit und ohne Fluoreszenzmarkierung. Für den Vergleich wurde der kleinste momentan erhältliche Marker, die synthetische Aminosäure Propargyl-L-Lysine (PRK), gewählt und in die kodierende Sequenz eines Zielproteins eingebaut. Die Visualisierung des Proteins erforderte die Anheftung einer synthetischen, fluoreszierenden Gruppe durch “Click-Chemie”. Dabei handelt es sich um eine sehr schnelle und selektiv organische Reaktion, die als Standardtechnik zur Markierung und Modifizierung von Biomolekülen etabliert ist.
Mittels super-auflösender Mikroskopie-Verfahren, der Ground-State Depletion Individual Molecule Return (GSDIM) Mikroskopie sowie der Stimulated Emission Depletion (STED) Mikroskopie, verglichen die Wissenschaftler anschließend die Organisation der markierten und unmarkierten Zielproteine in Zellen. Lediglich bei sechs der 26 untersuchten Proteine ließ sich eine leichte Beeinträchtigung der Proteinorganisation erkennen. In allen anderen Fällen bildeten die Proteine mit und ohne Fluoreszenzmarkierung vergleichbare molekulare Strukturen. Insgesamt bewerteten die Forscher daher die von ihnen gewählte Markierung als taugliches Instrument für die Nanoskopie.
„Auch andere Forschergruppen, die generell Probleme mit der Fluoreszenzmarkierung ihrer Proteine haben, sollten ausprobieren, ob unser Marker eine effiziente Alternative ist”, sagt Erstautorin Dr. Ingrid Vreja vom Institut für Neuro- und Sin-nesphysiologie der UMG. Mit der Publikation ihrer Studie stellen die Wissenschaftler ein detailliertes Protokoll zur Verfügung, das es anderen Wissenschaftlern ermöglicht, das beschriebene Verfahren für die Markierung der eigenen Zielproteine anzuwenden.
Quelle: Exzellenzcluster und DFG-Forschungszentrum Mikroskopie im Nanometerbereich und Molekularphysiologie des Gehirns
01.02.2016


