Supercomputer
Durchbruch für personalisierte Medizin dank SuperMUC
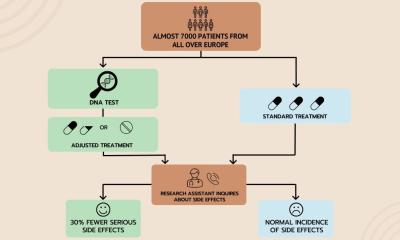
Einem europäischen Forscherteam gelang unter Leitung von Professor Peter Coveney vom Centre for Computational Science am University College London (UCL) ein großartiger Erfolg auf dem Gebiet der personalisierten Medizin: Peter Coveney und sein Team konnten zeigen, dass es möglich ist, anhand von Genom-Daten von Patientinnen mithilfe des Supercomputers zu erkennen, welche Standardmedikamente gegen Brustkrebs bei diesen Patientinnen nicht helfen können.

Dem Team stand der gesamte Höchstleistungsrechner SuperMUC am Leibniz-Rechenzentrum (LRZ) der Bayerischen Akademie der Wissenschaften mit allen seinen Ressourcen im Rahmen eines „Extreme Scaling Workshops“ zur alleinigen Nutzung zur Verfügung.
Wenn eine Patientin an Brustkrebs leidet, wird der Arzt ihr zuerst eines der üblichen Standardmedikamente verschreiben, es sei denn, er wüsste vorher, dass es bei ihr nicht wirken kann. Diese Unwirksamkeit kann man vorab erkennen, indem man das Genom untersucht. Basierend auf dieser Genom-Untersuchung kann der Arzt an-schließend das jeweils wirksame Medikament wählen. Die Medizin wird also exakt auf die Person abgestimmt. Die Möglichkeiten dieser personalisierten Medizin werden dank der wissenschaftlichen Fortschritte der letzten Jahrzehnte immer besser. Dazu tragen auch Supercomputer bei.
Peter Coveney ist Professor für physikalische Chemie. Er untersucht, warum zwei der üblichen Medikamente bei Patientinnen mit bestimmten Veränderungen im Erbmaterial nicht wirken. Coveney kennt das Protein, mit dem die Medikamente in Wechselwirkung treten. Und er kann berechnen, wie stark diese Wechselwirkung ist. Davon hängt es ab, ob die Medikamente heilen können oder nicht. Die Mutationen im Genom ändern die Aminosäuren im Protein. Dadurch ändert sich die Struktur des Proteins und die Wechselwirkung des Proteins mit dem Medikament. Diese Wechselwirkungen kann Peter Coveney auf dem SuperMUC sehr genau berechnen.
Diese Rechnungen beruhen auf unseren genauen Kenntnissen der Moleküle, die in vielen Jahrzehnten experimenteller und theoretischer Forschung von Wissenschaftle-rinnen und Wissenschaftlern zusammengetragen wurden. Der heutige Kenntnisstand ist so detailliert, dass man heute sehr genaue Aussagen machen kann, wenn man die gigantischen Rechenkapazitäten zur Verfügung hat, die dafür nötig sind.
Solche Rechenkapazitäten standen dem Team für das vergangene Wochenende zur Verfügung. Das LRZ bietet Spitzenforschern die Möglichkeit, den gesamten Höchst-leistungsrechner SuperMUC mit seinen fast 250.000 Rechenkernen im Rahmen der „Extreme Scaling Workshops“ für ausgewählte Simulationen exklusiv zu nutzen. Ins-gesamt über 6 Petaflops, also mehr als 6 Billiarden Rechenoperationen pro Sekunde, standen Peter Coveneys Team von Samstagnachmittag bis Montagmorgen zur Verfü-gung. 37 Stunden liefen ihre Rechnungen. 5.000 Gigabyte Ergebnisdaten wurden da-bei erzeugt. Vergleicht man diese Rechenleistung mit herkömmlichen PCs, so hätten 250.000 Menschen parallel 37 Stunden lang an Standard-PCs rechnen müssen.
„Wir haben alle unsere Ziele und mehr erreicht, weil die Rechnungen viel schneller liefen, als wir erwartet hatten. Eine erste Analyse zeigt, dass die Simulationen neue Ein-sichten liefern werden, wie die zwei häufigsten Mutationen die Resistenz gegen die wichtigsten Medikamente gegen Brustkrebs verursachen, indem sie die Bindung der Medikamente an das Protein verändern.“, äußerte sich Coveney am Montagmorgen begeistert. Allen Beteiligten war vorher bewusst, dass es eine große Herausforderung würde, diese Simulationen erfolgreich durchzuführen.
Mit ihren Rechnungen konnten sie nachweisen, dass es möglich ist, innerhalb weniger Stunden vorherzusagen, auf welche Weise ein bestimmtes Medikament an einer be-stimmten Stelle im Körper – in diesem Fall an einem bestimmten Protein – wirken wird, und zwar nicht nur für zwei Brustkrebs-Medikamente. „Wir haben insgesamt fünfzig Medikamente daraufhin untersucht, wie sie an verschiedene Proteine binden, um ihre Eignung für die Entwicklung und Klinische Anwendung neuer Medikamente bei verschiedenen Erkrankungen einordnen zu können.“, so Peter Coveney.
Die Forschungen finden im Rahmen des EU-geförderten Projektes „COMPAT“ statt, an dem mit dem University College London und dem LRZ insgesamt elf europäische Partner beteiligt sind. COMPATs Ziel ist es, den Einsatz von Simulationen für den Fortschritt von Wissenschaft und Technik an der Weltspitze voranzutreiben.
„Das Leibniz-Rechenzentrum ist stolz darauf, dass der vom Freistaat Bayern und dem Bund gemeinsam über das Gauss Centre for Supercomputing finanzierte SuperMUC auch für die personalisierte Medizin in seiner vollen Leistungsfähigkeit nutzbar ist.“, sagt Prof. Arndt Bode, Vorsitzender des Direktoriums des LRZ. „Die personalisierte Medizin hilft mit Unterstützung von Höchstleistungsrechnern Krankheiten zu heilen und dadurch das Leben der Menschen zu verbessern.“, erklärt Prof. Dieter Kranzlmüller, Direktor des LRZ und COMPAT-Projektpartner.
Quelle: Bayerische Akademie der Wissenschaften
17.06.2016